Background
Los cromosomas eucarióticos comúnmente tienen regiones de ADN satélite abundantes en repeticiones en tándem en cromatina centromérica y pericentromérica. La heterocromatina centromérica es bien conocida por establecer la función cinetocórica y permitir una segregación cromosómica fiel. Sin embargo, el papel del ADN satelital pericéntrico es menos comprendido, particularmente debido a la falta de codificación de proteínas y conservación en todas las especies. Estudios previos han mostrado roles para contextos específicos solamente, pero no se sabe si hay un papel más central para el ADN satelital eucariótico, especialmente dada su abundancia en el genoma.
Con este nuevo preprint, Jagannathan, Cummings y Yamashita dan seguimiento a su reciente estudio eLife que discute la formación y el papel de los cromocentros, o el agrupamiento de múltiples cromosomas, para mantener la organización nuclear. Abordan varias preguntas nuevas, incluyendo cómo todos los cromosomas de Drosophila melanogaster en un genoma pueden agruparse en cromocentros, y cómo múltiples ADN satelitales y sus correspondientes proteínas de unión al ADN pueden modular este proceso juntos.
Hallazgos clave
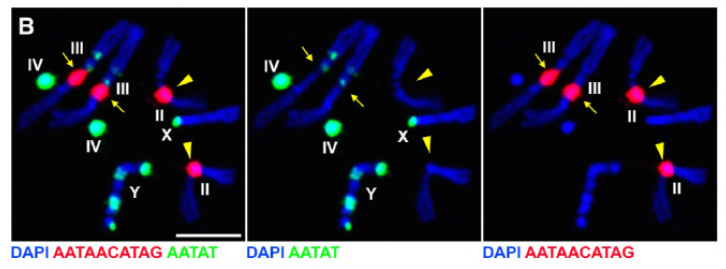
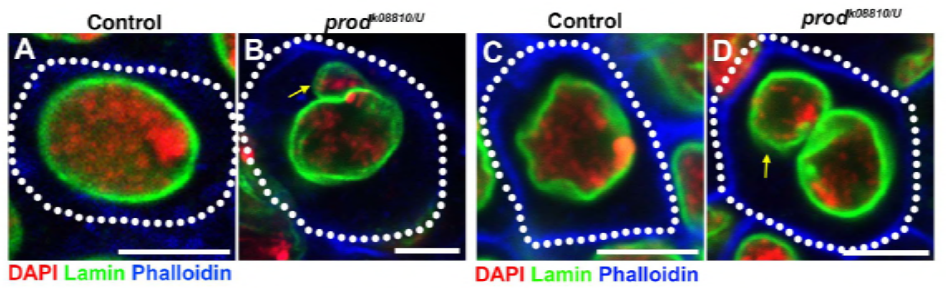
Dado que su trabajo anterior identificó una proteína, D1, que era responsable de agrupar los cromosomas X, Y y 4 en cromocentros, el grupo dirigió su atención a los cromosomas 2 y 3, los autosomas principales en D. melanogaster, para abordar cómo todos los cromosomas en el genoma pueden agruparse en cromocentros. Esto los llevó a descubrir una repetición de satélite más larga en los autosomas que estaba unida por la proteína Prod (disruptor de proliferación). La alteración de la función de prod llevó a la formación de micronúcleos y al aumento del daño al ADN, lo que en última instancia resultó en la muerte celular. Esto también se vio en su trabajo anterior con mutantes D1. Curiosamente, mientras que las mutaciones D1 afectaron la línea germinal, las mutaciones prod no lo hicieron, lo que sugiere que ambas actúan sobre diferentes tejidos a pesar de sus funciones similares de formación de cromocentro. Sin embargo, los mutantes dobles D1 prod no se desarrollan más allá de las etapas embrionarias y tienen un aumento de micronúcleos, lo que establece el requisito esencial de la formación del cromocentro a través del ADN satelital.
Prod claramente tiene un papel similar al de D1 en la formación de cromocentros. Los autores expresaron Prod ectópicamente en un tejido donde normalmente no está presente y causaron la formación de hilos de cromatina que establecen varios territorios cromosómicos que conectan los autosomas. Esto muestra claramente que Prod es suficiente para agrupar estos cromosomas a través de su unión al ADN satélite, lo que explica el mecanismo de cómo se forman los cromocentros.
En este punto, Jagannathan y Cummings et al han establecido que los cromosomas Prod y D1 se agrupan a través del ADN satélite. Pero dado que ambas proteínas actúan sobre diferentes conjuntos de cromosomas, ¿cómo se encapsula un conjunto de cromosomas de un genoma completo en un núcleo? Prod y D1 no parecían interactuar juntos a través de la inmunoprecipitación, lo que sugiere solo una interacción débil o transitoria. A través de imágenes en vivo, los autores descubrieron una interacción de «besar y correr»: los focos D1 y los focos Prod se tocan brevemente y luego se separan, lo que indica un proceso dinámico de formación del cromocentro. Ambas proteínas parecen ser mutuamente dependientes de la presencia funcional de la otra, ya que los mutantes prod mostraron una agrupación defectuosa de D1 en los núcleos y viceversa. Esta interdependencia proporciona una red para establecer la agrupación de todos los cromosomas en un genoma.
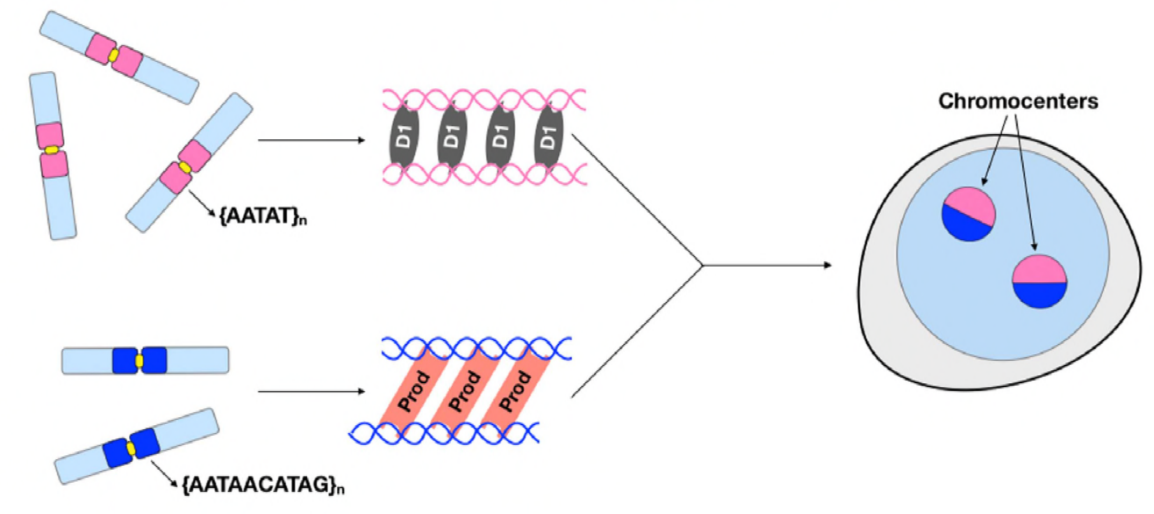
En resumen, Jagannathan y Cummings et al han demostrado que Prod y D1 crean una red donde ambas proteínas se unen a sus respectivas secuencias de ADN satélite para traer todos los cromosomas a un cromocentro, y finalmente empaquetar todo el genoma correctamente en el núcleo. Su estudio demuestra la importancia del ADN satelital, abordando no solo las consecuencias biológicas moleculares y celulares de las perturbaciones Prod y D1, sino también la importancia evolutiva de estas proteínas y del ADN satelital.
Preguntas para los autores
El estudio elife mostró que la pérdida de D1 hace que los micronúcleos se desprendan del núcleo principal, ¿causa la pérdida de prod la formación de micronúcleos de la misma manera?
¿La Prod y la D1 interactúan con otras proteínas aparte de sus interacciones transitorias de «besar y correr»? ¿Se sabe que estas proteínas funcionan en otros procesos relacionados con la integridad nuclear y/o el empaquetado del genoma?
En organismos con más de 4 cromosomas, ¿cuántas proteínas satélite de unión al ADN formadoras de cromocentros podrían ser necesarias para agrupar un genoma más grande con más cromosomas?
La discusión menciona que D. simulans no tiene el ADN satélite que se une a D. melanogaster Prod, entonces, ¿cuál es la proteína similar a D. simulans y cuán similar es a D. melanogaster en estructura y función?
Etiquetas: biología celular, drosophila, moscas de la fruta, genética, empaquetado del genoma, adn satelital
Publicado el: 11 de diciembre de 2018
doi: https://doi.org/10.1242/prelights.6160