Bakgrund
eukaryota kromosomer har vanligtvis regioner av satellit-DNA rikligt i tandemupprepningar vid centromeriskt och pericentromeriskt kromatin. Centromeriskt heterochromatin är välkänt för att etablera kinetochore-funktion och möjliggöra trogen kromosomsegregering. Emellertid är rollen av pericentriskt satellit-DNA mindre förstått, särskilt på grund av bristen på proteinkodning och bevarande över arter. Tidigare studier har visat Roller endast för specifika sammanhang, men huruvida det finns en mer central roll för eukaryot satellit-DNA, särskilt med tanke på dess överflöd i genomet, är inte känt.
med detta nya förtryck följer Jagannathan, Cummings och Yamashita upp sin senaste eLife-studie som diskuterar bildandet och rollen av kromocenter, eller buntning av flera kromosomer, för att upprätthålla kärnorganisation. De tar upp flera nya frågor, inklusive hur alla Drosophila melanogaster-kromosomer i ett Genom kan buntas i kromocenter, och hur flera satellit-DNA och deras motsvarande DNA-bindande proteiner kan modulera denna process tillsammans.
Nyckelfynd
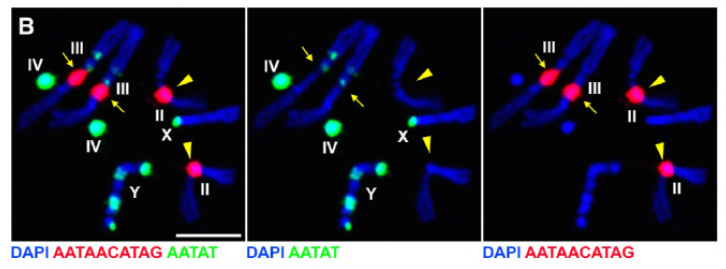
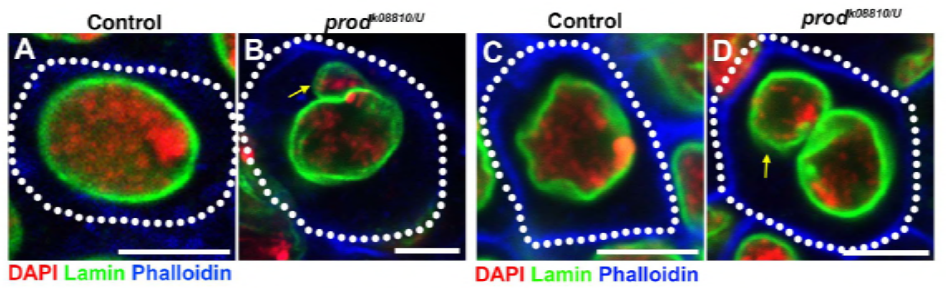
eftersom deras tidigare arbete identifierade ett protein, D1, som var ansvarigt för att kombinera kromosomer X, Y och 4 i kromocenter, vände gruppen sin uppmärksamhet mot kromosomer 2 och 3, de viktigaste autosomerna i D. melanogaster, för att ta itu med hur alla kromosomer i genomet kan grupperas i kromocenter. Detta ledde dem att upptäcka en längre satellitrepetition på autosomerna som var bundna av Prod (proliferation disrupter) – proteinet. Störande prod-funktion ledde till mikrokärnbildning och ökad DNA-skada, vilket slutligen resulterade i celldöd. Detta sågs också i deras tidigare arbete med D1-mutanter. Intressant, medan D1-mutationer påverkade könslinjen, gjorde prod-mutationer inte, vilket tyder på att båda verkar på olika vävnader trots deras liknande kromocenterbildande funktioner. D1-prod-dubbla mutanter misslyckas emellertid med att utvecklas förbi de embryonala stadierna och har ökat mikrokärnor, vilket fastställer det väsentliga kravet på kromocenterbildning via satellit-DNA.
Prod har tydligt en liknande roll som D1 för att bilda kromocenter. Författarna uttryckte Prod ektopiskt i en vävnad där den normalt inte är närvarande och orsakade bildandet av kromatintrådar som etablerar olika kromosomområden som förbinder autosomerna. Detta visar tydligt att Prod är tillräckligt för att bunta samman dessa kromosomer genom sin satellit-DNA-bindning, vilket förklarar mekanismen för hur kromocenter bildas.
vid denna tidpunkt har Jagannathan och Cummings et al fastställt att Prod-och D1-klusterkromosomer genom satellit-DNA. Men eftersom båda proteinerna verkar på olika uppsättningar kromosomer, hur är ett helt genoms uppsättning kromosomer inkapslade i en kärna? Prod och D1 verkade inte interagera tillsammans genom immunutfällning, vilket tyder på endast en svag eller övergående interaktion. Genom levande avbildning upptäckte författarna en” kiss and run ” – interaktion-D1-foci och Prod-foci berör kort och separerar sedan, vilket indikerar en dynamisk process för kromocenterbildning. Båda proteinerna verkar också vara ömsesidigt beroende av den andras funktionella närvaro, eftersom prod-mutanter visade defekt D1-kluster i kärnor och vice versa. Detta ömsesidiga beroende ger ett nätverk för att upprätta buntning av alla kromosomer i ett genom.
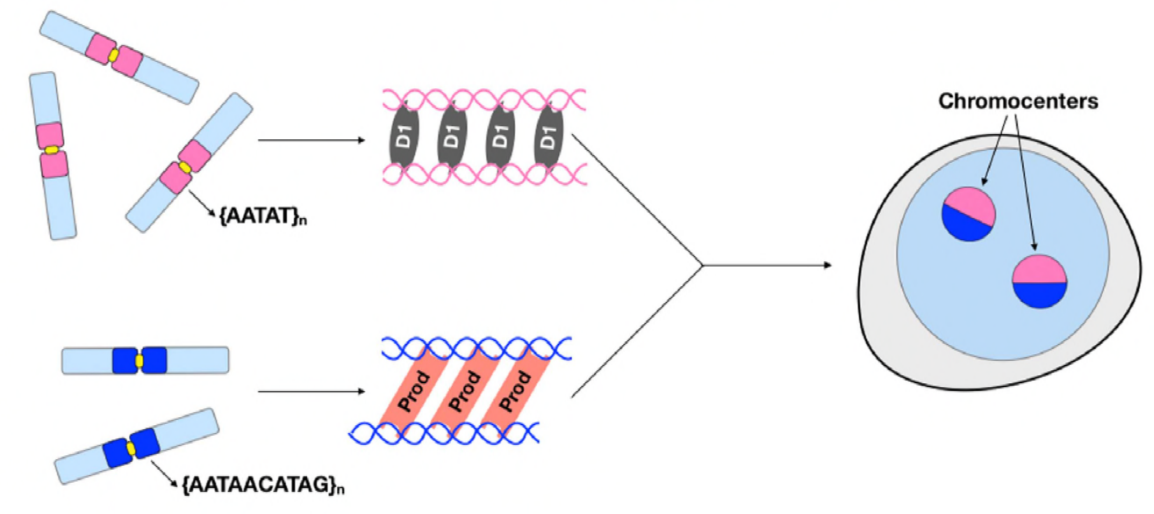
Sammanfattningsvis har Jagannathan och Cummings et al visat att Prod och D1 skapar ett nätverk där båda proteinerna binder sina respektive satellit-DNA-sekvenser för att bringa alla kromosomer i ett kromocenter och så småningom paketera hela genomet ordentligt i kärnan. Deras studie visar vikten av satellit-DNA, som inte bara behandlar de molekylära och cellbiologiska konsekvenserna av Prod-och D1-störningar utan också den evolutionära betydelsen av dessa proteiner och satellit-DNA också.
frågor till författarna
elife – studien visade att D1-förlust orsakar mikrokärnor att knoppa av från huvudkärnan-orsakar prod-förlust mikrokärnbildning på samma sätt?
interagerar Prod och D1 med andra proteiner förutom deras övergående ”kiss and run” – interaktioner? Är dessa proteiner kända för att fungera i andra processer relaterade till kärnintegritet och/eller genomförpackning?
i organismer med mer än 4 kromosomer, hur många kromocenterbildande, satellit-DNA-bindande proteiner kan vara nödvändiga för att bunta ett större genom med fler kromosomer?
diskussionen nämner att D. simulans inte har satellit-DNA som binder D. melanogaster Prod, så vad är det Prod-liknande proteinet i D. simulans och hur liknar det D. melanogaster i struktur och funktion?
taggar: cellbiologi, drosophila, fruktflugor, genetik, genom förpackning, satellit-dna
publiceras på: 11 December 2018
doi: https://doi.org/10.1242/prelights.6160