Hintergrund
Eukaryotische Chromosomen weisen häufig Regionen der Satelliten-DNA auf, die in Tandem-Wiederholungen bei zentromerem und perizentromerem Chromatin reichlich vorhanden sind. Es ist bekannt, dass zentromeres Heterochromatin die Kinetochorfunktion etabliert und eine getreue Chromosomensegregation ermöglicht. Die Rolle der perizentrischen Satelliten-DNA ist jedoch weniger bekannt, insbesondere aufgrund des Mangels an Proteinkodierung und -konservierung über Arten hinweg. Frühere Studien haben nur Rollen für bestimmte Kontexte gezeigt, aber ob es eine zentralere Rolle für eukaryotische Satelliten-DNA gibt, insbesondere angesichts ihrer Häufigkeit im Genom, ist nicht bekannt.
Mit diesem neuen Preprint knüpfen Jagannathan, Cummings und Yamashita an ihre jüngste eLife-Studie an, in der die Bildung und Rolle von Chromozentren oder die Bündelung mehrerer Chromosomen zur Aufrechterhaltung der nuklearen Organisation diskutiert wurden. Sie befassen sich mit mehreren neuen Fragen, darunter, wie alle Drosophila melanogaster-Chromosomen in einem Genom in Chromozentren gebündelt werden können und wie mehrere Satelliten-DNAs und ihre entsprechenden DNA-Bindungsproteine diesen Prozess gemeinsam modulieren können.
Schlüsselergebnisse
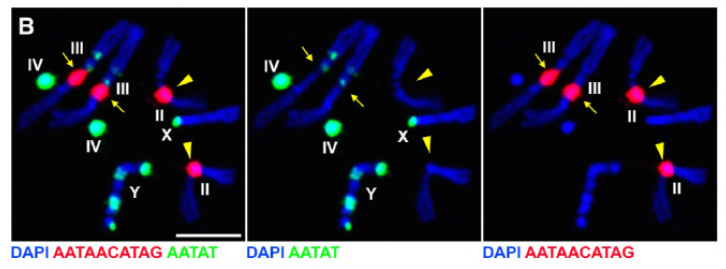
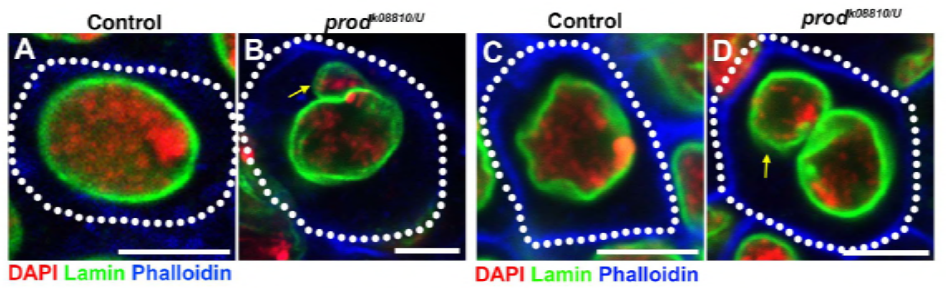
Da ihre früheren Arbeiten ein Protein, D1, identifizierten, das für die Bündelung der Chromosomen X, Y und 4 in Chromozentren verantwortlich war, wandte sich die Gruppe den Chromosomen 2 und 3 zu, den wichtigsten Autosomen in D. melanogaster, um zu untersuchen, wie alle Chromosomen im Genom in Chromozentren gruppiert werden können. Dies führte dazu, dass sie eine längere Satellitenwiederholung auf den Autosomen entdeckten, die durch das Prod-Protein (Proliferation Disruptor) gebunden war. Die Störung der Prod-Funktion führte zur Bildung von Mikronukleien und erhöhten DNA-Schäden, was letztendlich zum Zelltod führte. Dies wurde auch in ihrer früheren Arbeit mit D1-Mutanten gesehen. Interessanterweise beeinflussten D1-Mutationen zwar die Keimbahn, Prod-Mutationen jedoch nicht, was darauf hindeutet, dass beide trotz ihrer ähnlichen chromozentrischen Funktionen auf unterschiedliche Gewebe einwirken. D1-prod-Doppelmutanten entwickeln sich jedoch nicht über das embryonale Stadium hinaus und weisen erhöhte Mikronuclei auf, wodurch die wesentliche Voraussetzung für die chromozentrische Bildung über Satelliten-DNA geschaffen wird.
Prod hat eindeutig eine ähnliche Rolle wie D1 bei der Bildung von Chromozentren. Die Autoren exprimierten Prod ektopisch in einem Gewebe, in dem es normalerweise nicht vorhanden ist, und verursachten die Bildung von Chromatinfäden, die verschiedene Chromosomengebiete etablieren, die die Autosomen verbinden. Dies zeigt deutlich, dass Prod ausreicht, um diese Chromosomen durch seine Satelliten-DNA-Bindung zu bündeln, was den Mechanismus der Bildung von Chromozentren erklärt.
Zu diesem Zeitpunkt haben Jagannathan und Cummings et al. festgestellt, dass Prod- und D1-Cluster-Chromosomen durch Satelliten-DNA. Aber da beide Proteine auf unterschiedliche Chromosomensätze einwirken, wie ist der Chromosomensatz eines gesamten Genoms in einem Kern eingekapselt? Prod und D1 schienen nicht durch Immunpräzipitation miteinander zu interagieren, was nur auf eine schwache oder vorübergehende Wechselwirkung hindeutet. Durch Live-Imaging entdeckten die Autoren eine „Kiss and Run“ -Interaktion – D1-Herde und Prod-Herde berühren sich kurz und trennen sich dann, was auf einen dynamischen Prozess der Chromozentrumsbildung hinweist. Beide Proteine scheinen auch gegenseitig von der funktionellen Präsenz des anderen abhängig zu sein, da Prod-Mutanten eine defekte D1-Clusterbildung in Kernen zeigten und umgekehrt. Diese Interdependenz bietet ein Netzwerk, um die Bündelung aller Chromosomen in einem Genom zu etablieren.
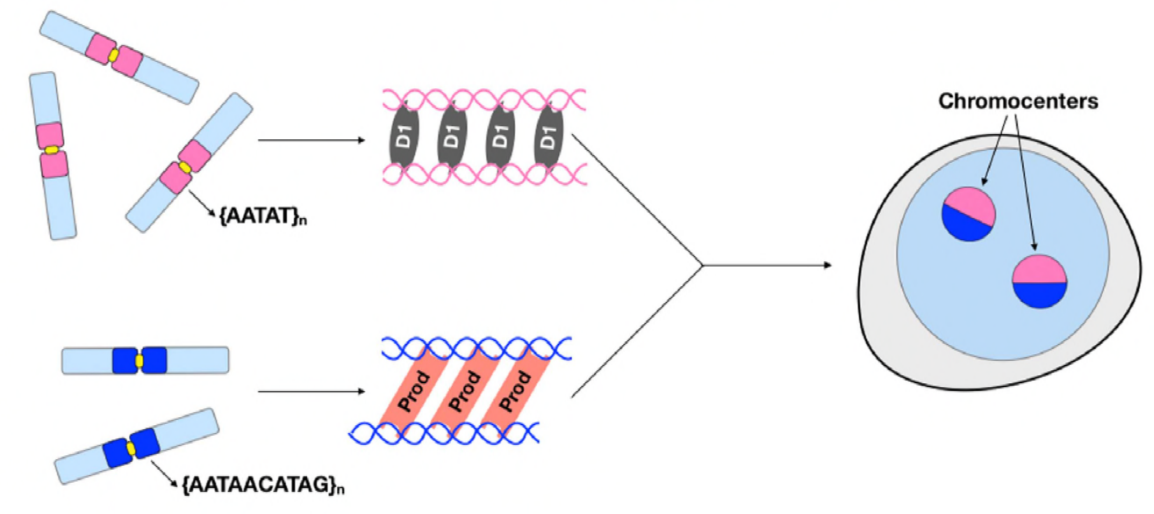
Zusammenfassend haben Jagannathan und Cummings et al. gezeigt, dass Prod und D1 ein Netzwerk bilden, in dem beide Proteine ihre jeweiligen Satelliten-DNA-Sequenzen binden, um alle Chromosomen in ein Chromozentrum zu bringen und schließlich das gesamte Genom richtig in den Kern zu verpacken. Ihre Studie zeigt die Bedeutung der Satelliten-DNA und befasst sich nicht nur mit den molekularen und zellbiologischen Konsequenzen von Prod- und D1-Störungen, sondern auch mit der evolutionären Bedeutung dieser Proteine und der Satelliten-DNA.
Fragen an die Autoren
Die elife-Studie zeigte, dass der D1-Verlust dazu führt, dass sich Mikronuklei vom Hauptkern ablösen – verursacht der Prod-Verlust auf die gleiche Weise die Bildung von Mikronuklei?
Interagieren Prod und D1 mit anderen Proteinen, abgesehen von ihren vorübergehenden „Kiss and Run“ -Interaktionen? Ist bekannt, dass diese Proteine in anderen Prozessen im Zusammenhang mit der nuklearen Integrität und / oder der Genomverpackung funktionieren?
In Organismen mit mehr als 4 Chromosomen, wie viele Chromozentrum bildende, Satelliten-DNA-bindende Proteine könnten notwendig sein, um ein größeres Genom mit mehr Chromosomen zu bündeln?
Die Diskussion erwähnt, dass D. simulans nicht die Satelliten-DNA hat, die D. melanogaster Prod bindet, also was ist das Prod-ähnliche Protein in D. simulans und wie ähnlich ist es D. melanogaster in Struktur und Funktion?
Schlagwörter: Zellbiologie, Drosophila, Fruchtfliegen, Genetik, Genomverpackung, Satelliten-DNA
Veröffentlicht am: 11.Dezember 2018
doi: https://doi.org/10.1242/prelights.6160