Contexte
Les chromosomes eucaryotes ont généralement des régions d’ADN satellite abondantes en répétitions en tandem à la chromatine centromérique et péricentromérique. L’hétérochromatine centromérique est bien connue pour établir la fonction kinétochore et permettre une ségrégation chromosomique fidèle. Cependant, le rôle de l’ADN satellite péricentrique est moins compris, en particulier en raison du manque de codage des protéines et de conservation d’une espèce à l’autre. Des études antérieures ont montré des rôles pour des contextes spécifiques seulement, mais on ignore s’il y a un rôle plus central pour l’ADN satellite eucaryote, en particulier compte tenu de son abondance dans le génome.
Avec cette nouvelle préimpression, Jagannathan, Cummings et Yamashita poursuivent leur récente étude eLife sur la formation et le rôle des chromocentres, ou le regroupement de chromosomes multiples, pour maintenir l’organisation nucléaire. Ils abordent plusieurs nouvelles questions, notamment comment tous les chromosomes de Drosophila melanogaster d’un génome peuvent être regroupés dans des chromocentres, et comment plusieurs ADN satellites et leurs protéines de liaison à l’ADN correspondantes peuvent moduler ce processus ensemble.
Résultats clés
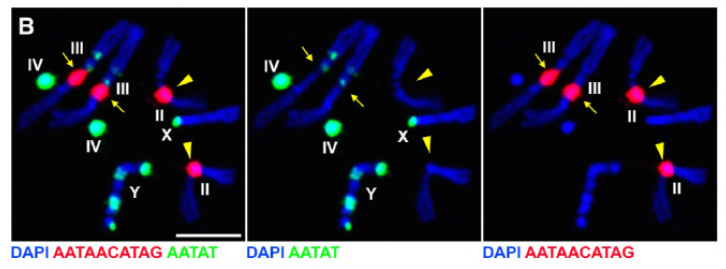
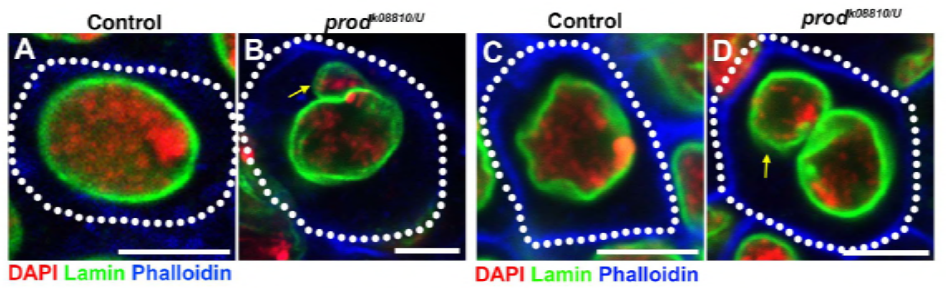
Depuis que leurs travaux antérieurs ont identifié une protéine, D1, responsable du regroupement des chromosomes X, Y et 4 en chromocentres, le groupe s’est tourné vers les chromosomes 2 et 3, les principaux autosomes de D. melanogaster, afin de déterminer comment tous les chromosomes du génome peuvent être regroupés en chromocentres. Cela les a amenés à découvrir une répétition satellite plus longue sur les autosomes qui était liée par la protéine Prod (proliferation disrupter). La perturbation de la fonction de la prod a conduit à la formation de micronoyaux et à une augmentation des dommages à l’ADN, entraînant finalement la mort cellulaire. Cela a également été vu dans leurs travaux précédents avec les mutants D1. Fait intéressant, alors que les mutations D1 affectaient la lignée germinale, les mutations prod ne l’ont pas fait, suggérant ainsi que les deux agissent sur des tissus différents malgré leurs fonctions similaires de formation de chromocentres. Cependant, les mutants doubles de la prod D1 ne parviennent pas à se développer au-delà des stades embryonnaires et ont augmenté les micronoyaux, établissant l’exigence essentielle de formation de chromocentres via l’ADN satellite.
Prod a clairement un rôle similaire à D1 dans la formation des chromocentres. Les auteurs ont exprimé la Prod de manière ectopique dans un tissu où elle n’est normalement pas présente et ont provoqué la formation de fils de chromatine qui établissent divers territoires chromosomiques reliant les autosomes. Cela montre clairement que la Prod est suffisante pour regrouper ces chromosomes ensemble grâce à sa liaison à l’ADN satellite, expliquant le mécanisme de formation des chromocentres.
À ce stade, Jagannathan et Cummings et al ont établi que Prod et D1 regroupent les chromosomes grâce à l’ADN satellite. Mais puisque les deux protéines agissent sur différents ensembles de chromosomes, comment l’ensemble des chromosomes d’un génome entier est-il encapsulé dans un noyau? Prod et D1 ne semblaient pas interagir ensemble par immunoprécipitation, suggérant seulement une interaction faible ou transitoire. Grâce à l’imagerie en direct, les auteurs ont découvert une interaction « baiser et courir » – les foyers D1 et les foyers Prod se touchent brièvement puis se séparent, indiquant un processus dynamique de formation de chromocentres. Les deux protéines semblent également dépendre mutuellement de la présence fonctionnelle de l’autre, car les mutants prod ont montré un groupement D1 défectueux dans les noyaux et vice versa. Cette interdépendance fournit un réseau pour établir le regroupement de tous les chromosomes dans un génome.
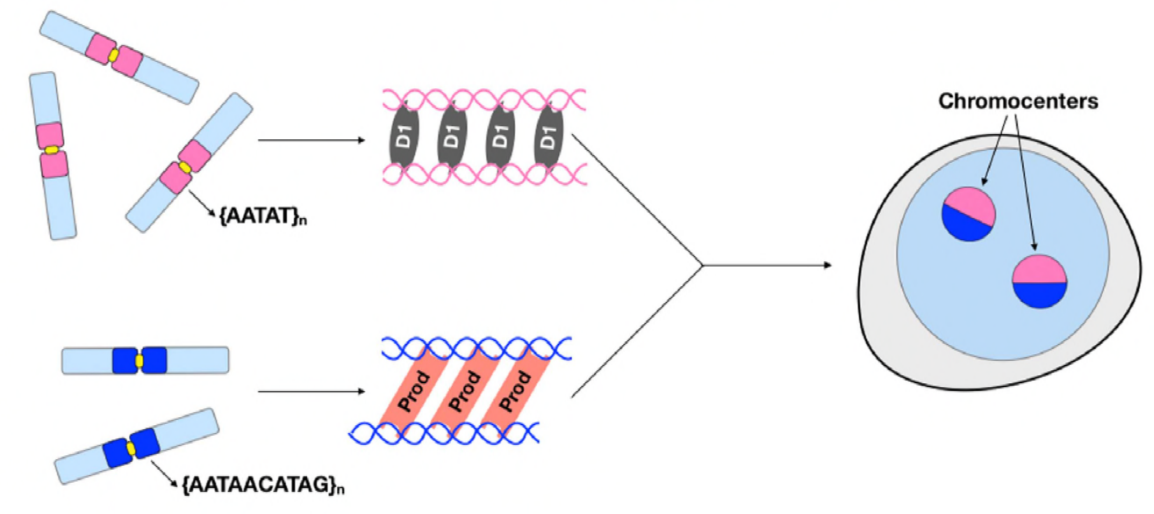
En résumé, Jagannathan et Cummings et al ont démontré que Prod et D1 créent un réseau dans lequel les deux protéines se lient à leurs séquences d’ADN satellites respectives afin d’amener tous les chromosomes dans un chromocentre, et éventuellement emballer correctement le génome entier dans le noyau. Leur étude démontre l’importance de l’ADN satellite, en abordant non seulement les conséquences biologiques moléculaires et cellulaires des perturbations de la Prod et de la D1, mais également la signification évolutive de ces protéines et de l’ADN satellite.
Questions pour les auteurs
L’étude elife a montré que la perte de D1 provoque le bourgeonnement des micronoyaux du noyau principal – la perte de prod provoque-t-elle la formation de micronoyaux de la même manière?
Prod et D1 interagissent-ils avec d’autres protéines en dehors de leurs interactions transitoires « baiser et courir »? Ces protéines sont-elles connues pour fonctionner dans d’autres processus liés à l’intégrité nucléaire et/ou au conditionnement du génome?
Dans les organismes comptant plus de 4 chromosomes, combien de protéines satellites se liant à l’ADN formant des chromocentres pourraient être nécessaires pour regrouper un génome plus grand avec plus de chromosomes?
La discussion mentionne que D. simulans n’a pas l’ADN satellite qui lie D. melanogaster Prod, alors quelle est la protéine de type Prod chez D. simulans et dans quelle mesure est-elle similaire à D. melanogaster en structure et en fonction?
Tags: biologie cellulaire, drosophile, mouches des fruits, génétique, emballage du génome, adn satellite
Posté le: 11 décembre 2018
doi: https://doi.org/10.1242/prelights.6160