fundo
cromossomas eucarióticos geralmente têm regiões de ADN satélite abundantes em repetições tandem na cromatina centromérica e pericentromérica. A heterocromatina centromérica é bem conhecida por estabelecer a função cinetocórea e permitir uma segregação cromossômica fiel. No entanto, o papel do ADN satélite pericêntrico é menos compreendido, particularmente devido à falta de codificação e conservação de proteínas entre as espécies. Estudos anteriores têm mostrado papéis apenas para contextos específicos, mas se há um papel mais central para o DNA de satélite eucariótico, especialmente dada a sua abundância no genoma, não é conhecido.
com este novo preprint, Jaganathan, Cummings, e Yamashita seguimento de seu estudo eLife recente discutindo a formação e o papel dos cromocentros, ou o agrupamento de vários cromossomos, para manter a organização nuclear. Eles abordam várias questões novas, incluindo como todos os cromossomos Drosophila melanogaster em um genoma podem ser agrupados em cromocentros, e como múltiplos DNAs satélite e suas correspondentes proteínas de ligação ao DNA podem modular este processo juntos.
principais conclusões
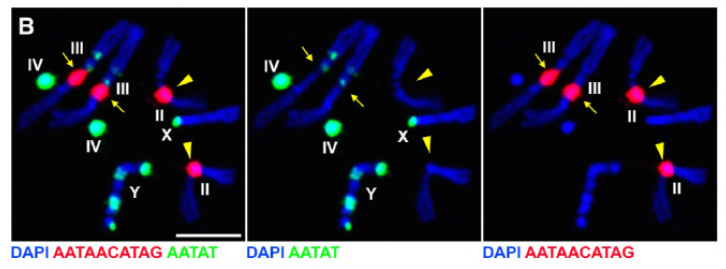
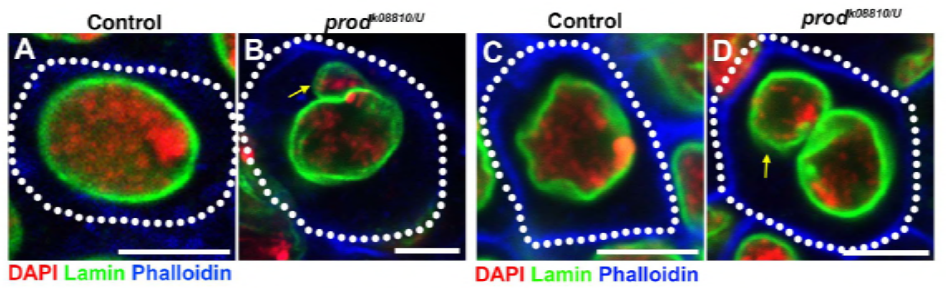
Desde seu trabalho anterior, identificaram uma proteína, D1, que foi o responsável pelo empacotamento de cromossomos X, Y, e 4 em chromocenters, o grupo se voltou sua atenção para os cromossomas 2 e 3, os principais autossomos em D. melanogaster, para o endereço como todos os cromossomos do genoma podem ser agrupados em chromocenters. Isto levou-os a descobrir uma repetição de satélite mais longa nos autossomas que estavam ligados pela proteína Prod (Disruptor de proliferação). O rompimento da função prod levou à formação de micronúcleos e aumento dos danos no DNA, resultando em morte celular. Isso também foi visto em seus trabalhos anteriores com mutantes D1. Curiosamente, enquanto as mutações D1 afetaram a germina, as mutações prod não o fizeram, sugerindo assim que ambos atuam em diferentes tecidos, apesar de suas funções semelhantes de formação de cromocentros. No entanto, os mutantes duplos D1 prod não se desenvolvem após os estágios embrionários e têm aumentado os micronúcleos, estabelecendo a exigência essencial de formação de cromocenter através do DNA satélite.
Prod claramente tem um papel semelhante ao D1 na formação de cromocentros. Os autores expressaram Prod ectopicamente em um tecido onde normalmente não está presente e causou a formação de fios de cromatina que estabelecem vários territórios cromossômicos conectando os autossomas. Isto mostra claramente que Prod é suficiente para agrupar esses cromossomos através de sua ligação de DNA satélite, explicando o mecanismo de como os cromocentros são formados.
neste ponto, Jaganathan e Cummings et al estabeleceram que cromossomas Prod e D1 agrupam-se através do DNA satélite. Mas como ambas as proteínas actuam em diferentes conjuntos de cromossomas, como é que um conjunto inteiro de cromossomas está encapsulado num núcleo? O Prod e o D1 não parecem interagir em conjunto através da imunoprecipitação, sugerindo apenas uma interacção fraca ou transitória. Através de imagens ao vivo, os autores descobriram um “kiss and run” interaction – D1 foci e Prod foci touch brevemente e, em seguida, separado, indicando um processo dinâmico de formação de cromocenter. Ambas as proteínas parecem ser mutuamente dependentes da presença funcional do outro também, uma vez que os mutantes prod mostraram clustering D1 defeituoso em núcleos e vice-versa. Esta interdependência fornece uma rede para estabelecer o agrupamento de todos os cromossomas num genoma.
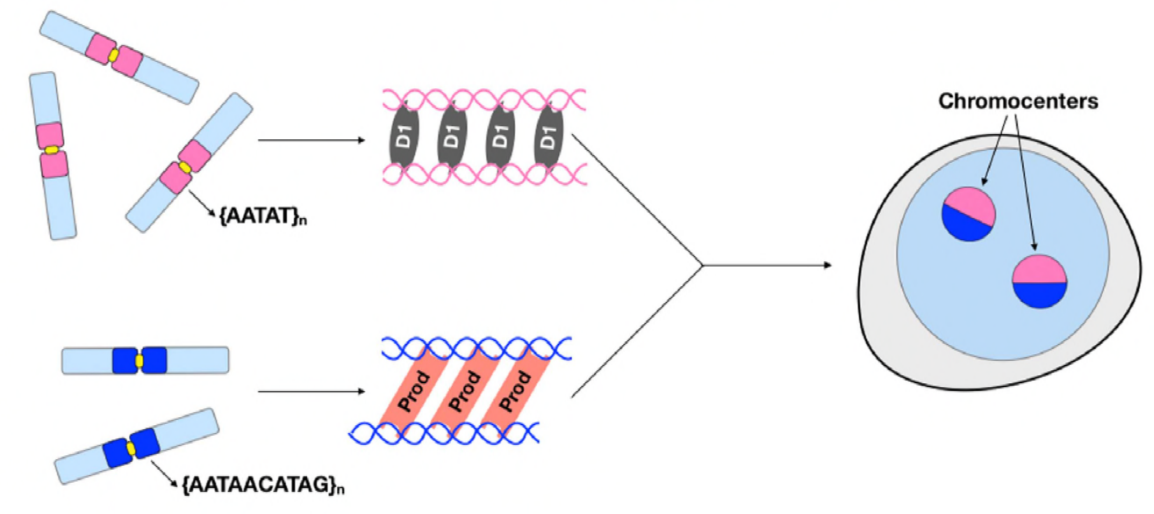
Em resumo, Jagannathan e Cummings et al demonstraram que, Prod e D1 criar uma rede onde ambas as proteínas ligam seus respectivos satélites sequências de DNA, a fim de trazer todos os cromossomos em um chromocenter, e, eventualmente, o pacote todo o genoma corretamente no núcleo. Seu estudo demonstra a importância do DNA satélite, abordando não só as consequências moleculares e celulares biológicas de perturbações de Prod e D1, mas também o significado evolutivo dessas proteínas e DNA satélite também.
perguntas para os autores
o estudo elife mostrou que a perda de D1 faz com que os micronúcleos brotem do núcleo principal – a perda de prod causa formação de micronúcleos da mesma forma?
do Prod e D1 interagem com outras proteínas para além das suas interacções transitórias de “kiss and run”? Sabe-se que estas proteínas funcionam noutros processos relacionados com a integridade nuclear e/ou a embalagem do genoma?
em organismos com mais de 4 cromossomas, quantas proteínas de ligação ao ADN por satélite, formadoras de cromocentros, podem ser necessárias para agrupar um genoma maior com mais cromossomas?
the discussion mentions that D. simulans doesn’t have the satellite DNA that binds D. melanogaster Prod, so what is the Prod-like protein in D. simulans and how similar is it to D. melanogaster in structure and function?
Tags: cell biology, drosophila, fruit flies, genetics, genome packaging, satellite dna
Posted on: 11 December 2018
doi: https://doi.org/10.1242/prelights.6160