Background
I cromosomi eucariotici hanno comunemente regioni di DNA satellitare abbondanti in ripetizioni tandem a cromatina centromerica e pericentromerica. L’eterocromatina centromerica è ben nota per stabilire la funzione cinetocore e consentire la segregazione cromosomica fedele. Tuttavia, il ruolo del DNA satellite pericentrico è meno compreso, in particolare a causa della mancanza di codifica e conservazione delle proteine tra le specie. Studi precedenti hanno dimostrato ruoli solo per contesti specifici, ma se esiste un ruolo più centrale per il DNA satellite eucariotico, soprattutto data la sua abbondanza nel genoma, non è noto.
Con questa nuova preprint, Jagannathan, Cummings e Yamashita seguono il loro recente studio eLife discutendo la formazione e il ruolo dei cromocentri, o il raggruppamento di cromosomi multipli, per mantenere l’organizzazione nucleare. Affrontano diverse nuove domande, tra cui come tutti i cromosomi Drosophila melanogaster in un genoma possono essere raggruppati in cromocentri e come più DNA satellitari e le loro corrispondenti proteine leganti il DNA possono modulare questo processo insieme.
risultati Chiave
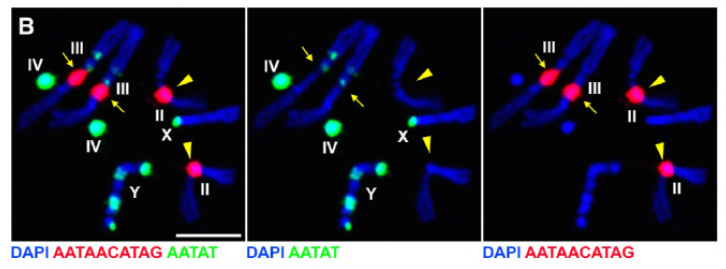
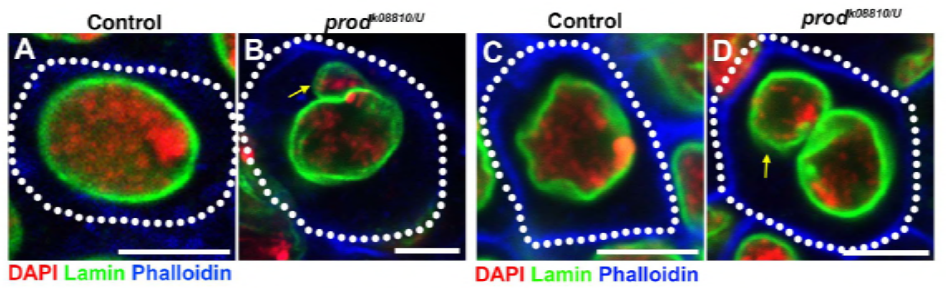
Dal loro lavoro precedente identificato una proteina D1, che è stato responsabile per impacchettare i cromosomi X, Y, e 4 in chromocenters, il gruppo ha rivolto la loro attenzione ai cromosomi 2 e 3, i principali autosomi in D. melanogaster, per affrontare come tutti i cromosomi del genoma che possono essere raggruppati in chromocenters. Ciò li ha portati a scoprire una ripetizione satellitare più lunga sugli autosomi che era legata dalla proteina Prod (proliferation disrupter). L’interruzione della funzione prod ha portato alla formazione di micronuclei e ad un aumento del danno al DNA, con conseguente morte cellulare. Questo è stato visto anche nel loro precedente lavoro con i mutanti D1. È interessante notare che, mentre le mutazioni D1 influenzavano la linea germinale, le mutazioni prod non lo facevano, suggerendo così che entrambe agissero su tessuti diversi nonostante le loro simili funzioni di formazione del cromocentro. Tuttavia, i doppi mutanti D1 prod non riescono a svilupparsi oltre gli stadi embrionali e hanno aumentato i micronuclei, stabilendo il requisito essenziale della formazione del cromocentro attraverso il DNA satellitare.
Prod ha chiaramente un ruolo simile a D1 nella formazione di cromocentri. Gli autori hanno espresso Prod ectopicamente in un tessuto dove normalmente non è presente e ha causato la formazione di fili di cromatina che stabiliscono vari territori cromosomici che collegano gli autosomi. Questo dimostra chiaramente che Prod è sufficiente per raggruppare questi cromosomi insieme attraverso il suo legame del DNA satellite, spiegando il meccanismo di come si formano i cromocentri.
A questo punto, Jagannathan e Cummings et al hanno stabilito che Prod e D1 cluster cromosomi attraverso il DNA satellitare. Ma dal momento che entrambe le proteine agiscono su diversi set di cromosomi, come è l’insieme di cromosomi di un intero genoma incapsulato in un nucleo? Prod e D1 non sembrano interagire insieme attraverso l’immunoprecipitazione, suggerendo solo un’interazione debole o transitoria. Attraverso l’imaging dal vivo, gli autori hanno scoperto un’interazione “kiss and run”: i fuochi D1 e i fuochi Prod toccano brevemente e poi si separano, indicando un processo dinamico di formazione del cromocentro. Entrambe le proteine sembrano dipendere reciprocamente dalla presenza funzionale dell’altro, poiché i prod mutanti hanno mostrato un clustering D1 difettoso nei nuclei e viceversa. Questa interdipendenza fornisce una rete per stabilire il raggruppamento di tutti i cromosomi in un genoma.
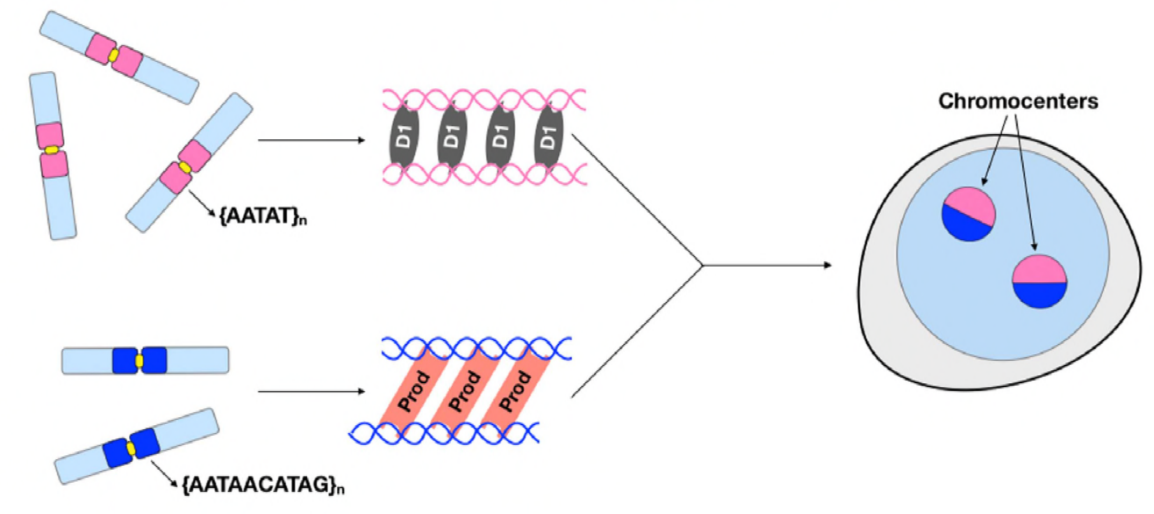
In sintesi, Jagannathan e Cummings et al hanno dimostrato che Prod e D1 creano una rete in cui entrambe le proteine legano le rispettive sequenze di DNA satellitare al fine di portare tutti i cromosomi in un cromocentro, e alla fine confezionare l’intero genoma correttamente nel nucleo. Il loro studio dimostra l’importanza del DNA satellitare, affrontando non solo le conseguenze biologiche molecolari e cellulari delle perturbazioni Prod e D1, ma anche il significato evolutivo di queste proteine e del DNA satellitare.
Domande per gli autori
Lo studio elife ha dimostrato che la perdita di D1 fa germogliare i micronuclei dal nucleo principale-la perdita di prod causa la formazione di micronuclei allo stesso modo?
Prod e D1 interagiscono con altre proteine oltre alle loro transitorie interazioni “kiss and run”? Queste proteine sono note per funzionare in altri processi legati all’integrità nucleare e / o al confezionamento del genoma?
Negli organismi con più di 4 cromosomi, quante proteine satellite leganti il DNA che formano il cromocentro potrebbero essere necessarie per raggruppare un genoma più grande con più cromosomi?
La discussione menziona che D. simulans non ha il DNA satellite che lega D. melanogaster Prod, quindi qual è la proteina Prod simile a D. simulans e quanto è simile a D. melanogaster nella struttura e nella funzione?
Tag: biologia cellulare, drosophila, moscerini della frutta, genetica, genome packaging, satellite dna
Pubblicato il: 11 dicembre 2018
doi: https://doi.org/10.1242/prelights.6160