Tło
chromosomy eukariotyczne często mają regiony DNA satelitarnego obfitujące w tandemowe powtórzenia w chromatynie centromerycznej i pericentromerycznej. Centromeryczna heterochromatyna jest dobrze znana z ustanowienia funkcji kinetochoru i umożliwienia wiernej segregacji chromosomów. Rola perycentrycznego DNA satelitarnego jest jednak mniej zrozumiała, szczególnie ze względu na brak kodowania białek i ochrony poszczególnych gatunków. Wcześniejsze badania wykazały rolę tylko dla konkretnych kontekstów, ale czy istnieje bardziej centralna rola dla eukariotycznego DNA satelitarnego, zwłaszcza biorąc pod uwagę jego obfitość w genomie, nie jest znane.
z tym nowym preprintem, Jagannathan, Cummings i Yamashita kontynuują swoje ostatnie badania eLife, omawiając tworzenie i rolę chromocentrów lub łączenie wielu chromosomów w celu utrzymania organizacji jądrowej. Odnoszą się one do kilku nowych pytań, w tym, jak wszystkie chromosomy Drosophila melanogaster w genomie mogą być wiązane w chromocentrach, i jak wiele satelitarnych DNA i odpowiadające im białka wiążące DNA mogą modulować ten proces razem.
kluczowe odkrycia
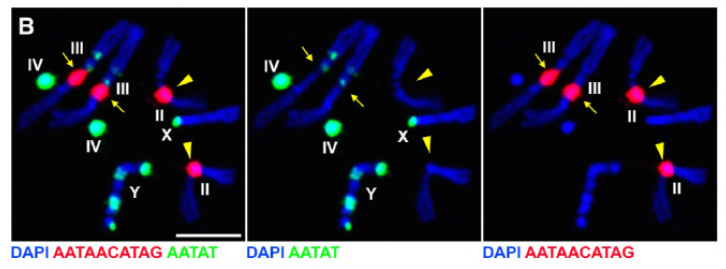
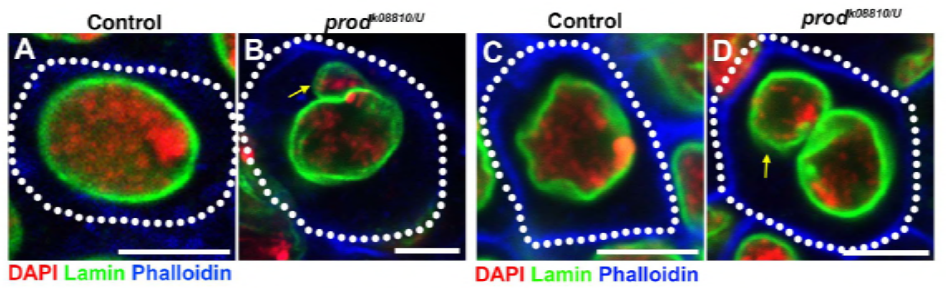
ponieważ ich poprzednia praca zidentyfikowała białko, D1, które było odpowiedzialne za łączenie chromosomów X, Y i 4 w chromocentry, grupa zwróciła swoją uwagę na chromosomy 2 i 3, główne autosomy w D. melanogaster, aby zająć się tym, jak wszystkie chromosomy w genomie mogą być zgrupowane w chromocentry. To doprowadziło ich do odkrycia dłuższego powtórzenia satelity na autosomach, które było związane z białkiem prod (proliferation disrupter). Zaburzenie funkcji prod doprowadziło do powstawania mikrojąder i zwiększonego uszkodzenia DNA, co ostatecznie doprowadziło do śmierci komórki. Było to również widoczne w ich wcześniejszej pracy z mutantami D1. Co ciekawe, podczas gdy mutacje D1 wpływały na linię płciową, mutacje prod nie, co sugeruje, że oba działają na różne tkanki pomimo ich podobnych funkcji chromocentrowych. Jednak podwójne mutanty D1 prod nie rozwijają się poza stadiami embrionalnymi i mają zwiększone mikrojądra, ustanawiając zasadniczy wymóg tworzenia chromocentrów za pomocą DNA satelitarnego.
prod wyraźnie ma podobną rolę do D1 w tworzeniu chromocentrów. Autorzy wyrazili Prodektopię w tkance, w której normalnie nie występuje i spowodowali powstanie nici chromatyny, które tworzą różne terytoria chromosomowe łączące autosomy. To wyraźnie pokazuje, że Prod jest wystarczający do wiązania tych chromosomów za pomocą satelitarnego wiązania DNA, wyjaśniając mechanizm powstawania chromocentrów.
w tym momencie Jagannathan i Cummings i wsp.ustalili, że chromosomy klastra Prod I D1 poprzez DNA satelitarne. Ale ponieważ oba białka działają na różne zestawy chromosomów, w jaki sposób cały zestaw chromosomów genomu jest zamknięty w jądrze? Wydaje się, że Prod I D1 nie oddziałują ze sobą poprzez immunoprecypitację, co sugeruje jedynie słabą lub przejściową interakcję. Poprzez obrazowanie na żywo autorzy odkryli interakcję” kiss and run ” – ogniska D1 i ogniska Prod dotykają krótko, a następnie oddzielają się, wskazując na dynamiczny proces tworzenia chromocentry. Oba białka wydają się być wzajemnie zależne od funkcjonalnej obecności drugiego, ponieważ mutanty prod wykazały wadliwe klastrowanie D1 w jądrach i odwrotnie. Ta współzależność zapewnia sieć do ustalenia wiązania wszystkich chromosomów w genomie.
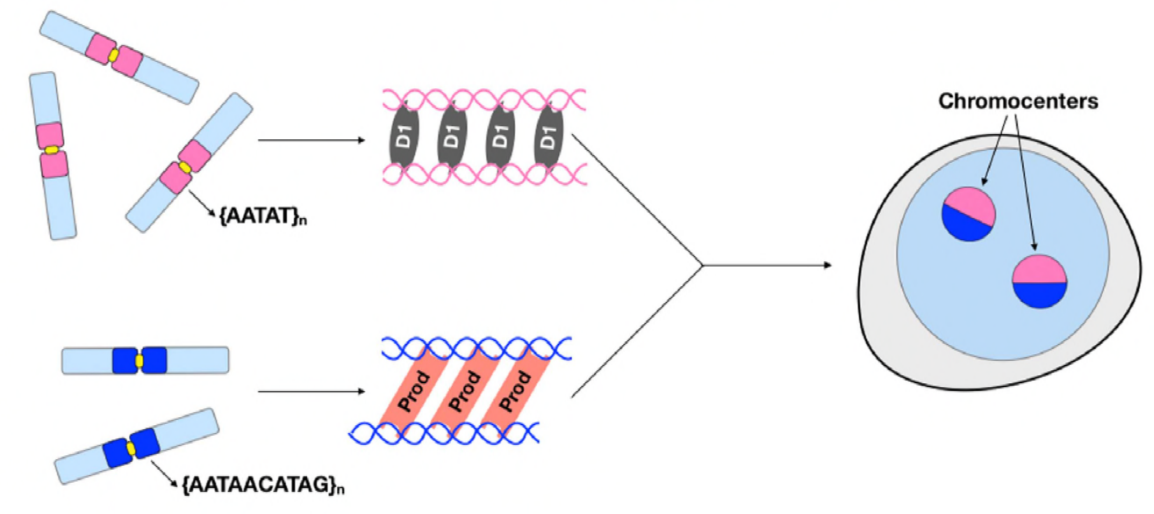
podsumowując, Jagannathan i Cummings i wsp.wykazali, że Prod I D1 tworzą sieć, w której oba białka wiążą swoje odpowiednie sekwencje DNA satelitarnego w celu przeniesienia wszystkich chromosomów do chromocentru i ostatecznie prawidłowo zapakować cały genom do jądra. Ich badania pokazują znaczenie satelitarnego DNA, zajmując się nie tylko molekularnymi i komórkowymi biologicznymi konsekwencjami perturbacji Prod I D1, ale także ewolucyjnym znaczeniem tych białek i satelitarnego DNA.
pytania do autorów
badanie elife wykazało, że utrata D1 powoduje oderwanie mikrojąder od głównego jądra – czy utrata prod powoduje tworzenie mikrojąder w ten sam sposób?
czy Prod I D1 oddziałują z innymi białkami poza ich przejściowymi oddziaływaniami „kiss and run”? Czy białka te działają w innych procesach związanych z integralnością jądrową i / lub pakowaniem genomu?
w organizmach z więcej niż 4 chromosomami, ile chromocentrowych, satelitarnych białek wiążących DNA może być potrzebnych do wiązania większego genomu z większą liczbą chromosomów?
dyskusja wspomina, że D. simulans nie ma satelitarnego DNA, które wiąże D. melanogaster Prod, więc jakie jest białko podobne do prod w D. simulans i jak podobne jest do D. melanogaster pod względem struktury i funkcji?
Tags: biologia komórki, drosophila, muszki owocowe, genetyka, opakowania genomu, DNA satelitarne
Posted on: 11th December 2018
doi: https://doi.org/10.1242/prelights.6160