Significância
Há um interesse crescente em circRNAs devido à sua implicação em muitos processos biológicos e doenças, além de seu potencial biomarcador. Eles são detectados principalmente pela presença de leituras mapeando sua junção backsplicing. No entanto, circRNAs não são mais as únicas transcrições que contêm tal junção, uma vez que estudos recentes revelaram que DNAs circulares são comuns e podem ser transcritas, resultando em transcrições que imitariam um sinal de circRNA. Portanto, este novo tipo de transcrição quimérica pode mudar a forma como a análise circRNA está sendo feita e impactar alguns dos resultados já relatados.
São RNAs circulares as únicas transcrições quiméricas?
RNA Circular (cirrnas) foram redescobertos há alguns anos como formas de RNA não-canonicamente esplicadas presentes em diferentes organismos, incluindo humanos (Salzman et al., 2012; Jeck et al., 2013). Eles são ligadas fechado transcrições formado através de um RNA volta de splicing do evento, onde uma emenda doador de um jusante exão junta-se a um montante de emenda acceptor levando ligadas fechado transcrições que são caracterizados pela presença de uma emenda de junção que faz circRNAs distinguíveis das suas linear homólogos (Figura 1A) (Zhang et al., 2016; Wilusz, 2018).
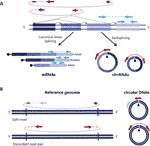
Figura 1 A Detecção de RNA circular e DNAs é baseada em leituras que abrangem a junção. A) a junção Backsplicada entre exões e exões canónicos é representada com linhas não contínuas e contínuas que dão origem, respectivamente, a circRNAs e mRNAs. Leituras emparelhadas abrangendo as junções backspliced são mostradas em vermelho e leituras emparelhadas consistentes com uma transcrição linear são mostradas em azul. B) à semelhança das circulares, as DNAs circulares (eccDNAs) são detectadas com base em variantes de leitura estrutural consistentes com um evento de circularização representado a vermelho .
Desde sua redescoberta, a comunidade científica tem atraído a sua atenção para circRNAs e investigou sua participação em diversos processos celulares na saúde e na doença (Haque e Harries, 2017), o seu potencial papel como biomarcadores (e Abu Jamal, de 2016), e suas funções reguladoras (Floris et al., 2016). As CircRNAs são hoje conhecidas por serem abundantes e estáveis no citosol e no núcleo (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) e também foram encontrados livres em biofluidas (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) e nas vesículas extracelulares (Kyoung Mi et al., 2017). O potencial do biomarcador de circRNAs foi intensamente estudado, de fato, foram publicados muitos estudos de caso-controle que procuram circRNAs de expressão diferente que poderiam ser biomarcadores de diferentes doenças. Até o momento, cirrnas tem sido implicada em várias doenças, incluindo câncer (Kristensen et al., 2017; Arnaiz et al., 2018), distúrbios neurológicos( Akhter, 2018), doenças cardiovasculares (Aufiero et al., 2019) e doenças imunes (Iparraguirre et al., 2017; Liu et al., 2019). Ao mesmo tempo, chegar a compreender plenamente a sua biogênese, características, funções e implicações na biologia humana permanecem como questões abertas para os pesquisadores no campo.Embora a função da maioria das circRNAs permaneça desconhecida, tem sido demonstrado que algumas circRNAs podem atuar como esponjas microRNAs, regulando os níveis de microRNA e sua atividade (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Eles estão envolvidos na regulação da expressão genética, regulando a transcrição de seus genes parentais, competindo com as proteínas lineares ou esponjas (Ashwal-Fluss et al., 2014; Li et al., 2018). Curiosamente, estudos de caracterização de ribossomas demonstraram recentemente que as circRNAs também podem ser traduzidas in vitro e in vivo (Legnini et al., 2017; Yang et al., 2017).
a principal característica das cirnas, e responsável pela maioria de suas propriedades especiais, é sua circularidade. Portanto, além de detectar sua junção esplicada traseira Característica, testando a circularidade dessas moléculas, é um dos pontos chave em cada estudo de circRNA. No entanto, muitos estudos têm baseado sua descoberta de cirnas em RNA total e podem, assim, ter interpretado algumas transcrições quiméricas lineares como circnas, resultando em deteções de circRNA falsas positivas. Para contornar este problema, a maioria dos estudos confirmaram a circularidade das transcrições encontradas pelo RNA-seq total usando RNase R, Northern blot ou métodos eletroforéticos (Jeck e Sharpless, 2014). No entanto, estas validações de circularidade também por vezes revelaram transcrições que parecem ser lineares, ao invés de circulares confirmando que a detecção de circnas a partir do RNA total pode levar a alguns falsos positivos. Estes falsos positivos foram atribuídos a artefatos técnicos ou transcrições derivados de eventos incomuns como duplicações de exon ou eventos de transplicação (Jeck e Sharpless, 2014; Szabo e Salzman, 2016). Dito isto, a opção de ter encontrado transcrições lineares verdadeiras, biologicamente ativas e funcionais que contêm uma sequência equivalente a uma junção backsplicing (a partir de agora chamada de transcrições lineares quiméricas), tem sido um pouco negligenciada porque uma fonte de tal ARN linear não tem sido conhecida por células saudáveis.
Circular de Dna como uma Fonte de Quiméricas Linear Transcrições
a Maioria do genoma humano está organizado em cromossomos lineares, no entanto, algumas exceções, tem sido aceito como o DNA mitocondrial, e aberrações cromossômicas, tais como DNA círculos de transporte de oncogenes (e.g. dobro de minutos) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) e cromossomas anelares (Tümer et al., 2004). Não foi até recentemente que DNAs circulares diferentes, como microDNAs (Shibata et al. Em 2012, a circular extrachromosómica DNAs (eccDNAs) também surgiu de grandes partes de diferentes genomas eucarióticos, incluindo humanos e leveduras (Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
DNAs circulares são formadas quando duas extremidades de um DNA linear são juntas, resultando em uma junção similar à junção backsplicada em circRNAs comumente chamada de junção breakpoint que é detectada com base em variantes de leitura estrutural consistentes com um evento de circularização (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (figura 1B). Eles geralmente variam de uma centena de bases a círculos megabase e podem conter exons e genes completos (Shibata et al ., 2012; Møller et al., 2015; Kumar et al., 2017; Turner et al., 2017; Møller et al., 2018) e enquanto algumas regiões do genoma são mais comumente encontrados no DNA circular (Sinclair e Guarente, 1997;; Møller et al., 2016; Turner et al., 2017; Møller et al., 2018), a maioria do DNA circular parece ocorrer aleatoriamente (Shibata et al., 2012; Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
curiosamente, em um recente artigo Møller et al. identificou milhares de eccDNAs em leucócitos e células musculares em controlos saudáveis. Com a idéia de investigar se eccDNAs poderia ser transcrito, uma biblioteca de mRNA também foi sequenciada a partir do tecido muscular e analisada para eventos de transcrição através da junção de breakpoint da eccdna detectada encontrando vários fósforos (Møller et al., 2018). Este achado sugere que o DNA circular no tecido saudável é transcrito, dando origem a transcrições lineares e poliadeniladas que carregarão uma sequência equivalente à sequência de retrosplicação de circRNAs (Møller et al., 2018) (Figura 2).
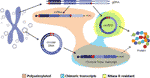
Figura 2 representação gráfica de diferentes transcrições a partir de ADN genómico ou ADN circular. Exons são coloridos em roxo e as junções backspliced ou junções quiméricas são mostradas em vermelho. As transcrições poliadeniladas, de junção quimérica contendo e resistentes à RNase R são destacadas em laranja, azul e amarelo, respectivamente.
a evidência transcritional de DNAs circulares, juntamente com sua abundância, nos leva a sugerir que DNAs circulares poderiam ser uma fonte natural de uma quantidade substancial de RNAs lineares Carregando junções quiméricas. Em muitos casos, estas junções quiméricas podem ser indistinguíveis das junções backsplicing das circRNAs, e, portanto, podem ser fatores de confusão em estudos circRNA. Nos parágrafos seguintes, explicaremos os dados que apoiam esta proposta.
detecção por circRNA: All That Glitters Is Not Gold
As previously introduced, circRNAs are formed though a non-canonical splicing event called backsplicing. Transcrições resultantes deste backsplicing evento tem um ligadas de circuito fechado estrutura nem 5′-3′ polaridade, nem um polyadenylated cauda e, mais importante, eles são caracterizados pela presença de um mexidos exão ordem relativa a transcrição linear (Zhang et al., 2016; Wilusz, 2018). Esta ordem de exon codificado torna-se evidente na junção traseira que liga uma sequência 5′ downstream com uma sequência 3′ upstream. Assim, todos os algoritmos de detecção de circRNA exploram a presença das junções de Spliced para trás como uma característica de diagnóstico para a identificação de circRNA (figura 1A).
diferentes métodos têm sido adaptados para a detecção destes cruzamentos. Arrays comerciais contendo sondas direcionadas para essas regiões com backspliced têm sido amplamente utilizados em estudos de rastreio de biomarcadores (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). A validação subsequente é muitas vezes também baseada na amplificação das junções backsplicing usando primers divergentes (Panda e Gorospe, 2018). Muitos outros trabalhos têm conduzido uma análise de sequenciamento de alto rendimento que supera uma das principais limitações dos arrays permitindo detectar não só as circRNAs anotadas, mas também eventos de circularização de RNA de novo de regiões genômicas onde nenhum circRNA foi anotado por estudos anteriores. Vários bioinformatic dutos têm sido desenvolvidos para a detecção de circRNAs no RNA-Seq conjuntos de dados, mas todos eles são baseados na presença de lê travessia sobre a volta de splicing cruzamentos e encontrar os mais confiável ainda é um desafio para bioinformatas (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
duas abordagens principais podem ser seguidas para a detecção de circRNAs em dados RNA-Seq. Em primeiro lugar, muitos estudos de ARN-seq com circRNA são baseados em amostras tratadas com RNase R, A fim de esgotar todas as RNAs lineares antes da sequenciação. Embora esta abordagem é especialmente concebido para os circRNA de detecção é de notar que RNase R degradação é variável, de que são raros os casos de RNase R resistentes linear RNAs e RNase R sensíveis circRNAs (Szabo e Salzman, de 2016) e que este tratamento requer uma alta de RNA de entrada que pode ser limitante para alguns tecidos. Outros estudos circRNA optam por sequenciar o RNA total, com depleção ribossómica (ribo -) ou não-poliadenilado (polyA -), onde podem ser encontrados RNAs lineares e circulares (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak et al., 2015). Esta abordagem evita o uso de RNase R, O que reduz a quantidade de RNA necessária para a sequenciação e permite estudar a expressão de outros tipos de RNAs a partir do mesmo conjunto de dados. It has been demonstrated that with a good sequencing depth and quality and a carefully data-analysis, true circRNAs can be detected from total RNA sequencing (Wang et al., 2017), no entanto, nesta segunda abordagem, é necessária uma confirmação de circularidade posterior para as circulares.
With the discovery of linear chimeric RNAs transcribed from circular DNAs, circRNAs are no longer the only transcripts with chimeric junctions. Portanto, é de extrema importância a observação de que, enquanto a primeira abordagem significativamente irão enriquecer o RNA da amostra em circRNAs de modo que a maioria dos detectado quimérica, uma junção correspondem a verdadeiras circRNAs, o segundo pode superestimar o número de circRNA transcrições atribuindo ao circRNAs o sinal de ambos os circRNAs e linear quimérica transcrições transcrita de Dna circular. Consequentemente, tendo em conta a coexistência de circRNAs e transcrições quiméricas lineares, a necessidade de testes de circularidade e testes de funcionalidade ganha importância e deve ser tido especial cuidado não só em métodos experimentais, mas também computacionais para evitar confundir transcrições quiméricas de DNAs circulares com circulares formadas por voltagem.
Discussão
O circRNA campo está ainda numa fase inicial, no entanto, circRNAs, já demonstrou ser surpreendente moléculas, envolvidos em muitos processos, com um grande potencial biomarcador e que também pode alterar a maneira como entendemos a transcrição e no processo de tradução. Por estas razões, eles estão ganhando atenção e o campo circRNA é, no momento, um dos campos de pesquisa de RNA mais ativos. No entanto, ainda há muitos conflitos, controvérsias e questões em aberto (Li, 2019) que precisam ser discutidos.
neste relatório, e na luz dos recentes avanços no DNA circular de campo, queremos ressaltar a transcrição de extrachromosomal DNA circular, como uma das principais fontes naturais de linear transcrições com emendados sinais de que poderiam estar interferindo com circRNA de dados (Møller et al., 2018). A partir de agora, além dos artefatos técnicos, duplicações e eventos de transplicação que poderiam levar a falsos positivos na detecção de circRNA, devemos levar também em conta a existência deste novo tipo de transcrições quiméricas. Por conseguinte, os ensaios de circularização e os ensaios funcionais são mais importantes do que nunca.
em qualquer caso, estas transcrições lineares quiméricas não devem ser consideradas apenas como um mero factor de confusão para estudos de circRNA. Apesar das implicações técnicas para a caracterização de circRNA, a existência destas moléculas de RNA linear quimérica do tipo circRNA provenientes de eccDNAs adiciona um novo tipo de molécula à lista cada vez maior de RNAs e expande a nossa visão sobre a complexidade do transcriptoma e sua regulação. Além disso, estas moléculas lineares de RNA provenientes do eccDNA também poderiam apresentar funções semelhantes ao circRNA, incluindo as funções reguladoras ou o potencial a ser traduzido. Os produtos genéticos provenientes de transcrições eccDNA podem potencialmente contribuir para o fenótipo de células somáticas e tecidos, tal como referido na levedura (Gresham et al., 2010; Demeke et al., 2015). No entanto, neste campo nascente, mais dados e pesquisas são necessários para começar a arranhar a superfície do iceberg.
Autor Contributions
LI, IP-L, BR, and do wrote the paper.Este estudo foi financiado pelo Instituto de Salud Carlos III através do projecto “PI17/00189” (Co-financiado pelo Fundo Europeu de Desenvolvimento Regional/Fundo Social Europeu) “Investing in your future”). A IP-L e a BR foram apoiadas pelo Conselho dinamarquês de investigação independente, 6108-00171B e LI foram apoiadas pelo Departamento de Educação do Governo Basco .
conflito de interesses
os autores declaram que a pesquisa foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.