Significance
C’è un crescente interesse per le CIRCRNA a causa della loro implicazione in molti processi biologici e malattie oltre al loro potenziale biomarcatore. Sono rilevati principalmente dalla presenza di letture che mappano la loro giunzione di backsplicing. Tuttavia, i CIRCRNA non sono più gli unici trascritti che contengono tale giunzione poiché studi recenti hanno rivelato che i DNA circolari sono comuni e possono essere trascritti con conseguente trascrizione che imiterebbe un segnale circRNA. Pertanto, questo nuovo tipo di trascrizione chimerica può cambiare il modo in cui viene eseguita l’analisi circRNA e influire su alcuni dei risultati già riportati.
Gli RNA circolari sono le uniche trascrizioni chimeriche?
Gli RNA circolari (circRNAs) sono stati riscoperti alcuni anni fa come forme di RNA non canonicamente giuntate presenti in diversi organismi tra cui gli esseri umani (Salzman et al., 2012; Jeck et al., 2013). Sono trascritti covalentemente chiusi formati attraverso un evento di back-splicing dell’RNA, dove un donatore di giunzione di un esone a valle si unisce a un accettore di giunzione a monte che porta a trascritti covalentemente chiusi caratterizzati dalla presenza di una giunzione di back-splicing che rende i CIRCRNA distinguibili dalle loro controparti lineari (Figura 1A) (Zhang et al., 2016; Wilusz, 2018).
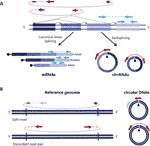
Figura 1 Il rilevamento di RNA e DNA circolari si basa su letture che coprono la giunzione. (A) L’unione Backspliced tra gli esoni e l’unione canonica degli esoni sono raffigurati con linee non continue e continue rispettivamente che danno origine a CIRCRNA e mRNA. Le letture di fine coppia che coprono le giunzioni backspliced sono mostrate in rosso e le letture di fine coppia coerenti con una trascrizione lineare sono mostrate in blu. (B) Analogamente ai CIRCRNA, i DNA circolari (ECCDNA) sono rilevati sulla base di varianti di lettura strutturale coerenti con un evento di circolarizzazione raffigurato in rosso .
Dalla loro riscoperta, la comunità scientifica ha attirato la sua attenzione sui CIRCRNA e ha studiato il loro coinvolgimento in diversi processi cellulari in salute e malattia (Haque e Harries, 2017), il loro potenziale ruolo di biomarcatori (Abu e Jamal, 2016) e le loro funzioni regolatorie (Floris et al., 2016). I CIRCRNA sono ora noti per essere abbondanti e stabili nel citosol e nel nucleo (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) e sono stati trovati anche liberi nei biofluidi (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) e nelle vescicole extracellulari (Kyoung Mi et al., 2017). Il potenziale biomarcatore di CIRCRNA è stato intensamente studiato, infatti, sono stati pubblicati molti studi di caso-controllo alla ricerca di CIRCRNA differenzialmente espressi che potrebbero essere biomarcatori di diverse malattie. Ad oggi, i CIRRNA sono stati implicati in diverse malattie tra cui il cancro (Kristensen et al., 2017; Arnaiz et al., 2018), disturbi neurologici (Akhter, 2018), malattie cardiovascolari (Aufiero et al., 2019) e malattie immuno-correlate (Iparraguirre et al., 2017; Liu et al., 2019). Allo stesso tempo, arrivare a comprendere appieno la loro biogenesi, le caratteristiche, le funzioni e le implicazioni nella biologia umana rimangono domande aperte per i ricercatori nel campo.
Sebbene la funzione della maggior parte dei CIRRNA rimanga sconosciuta, è stato dimostrato che alcuni CIRRNA possono agire come spugne microRNA, regolando i livelli di microRNA e la loro attività (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Sono coinvolti nella regolazione dell’espressione genica regolando la trascrizione dei loro geni parentali, in competizione con le proteine di splicing o spugnatura lineari (Ashwal-Fluss et al., 2014; Li et al., 2018). È interessante notare che gli studi di profilazione del ribosoma hanno recentemente dimostrato che i CIRCRNA possono anche essere tradotti sia in vitro che in vivo (Legnini et al., 2017; Yang et al., 2017).
La caratteristica principale dei CIRCRNA, e responsabile della maggior parte delle loro proprietà speciali, è la loro circolarità. Pertanto, oltre a rilevare la loro caratteristica giunzione back-spliced, testare la circolarità di queste molecole, è uno dei punti chiave in ogni studio circRNA. Tuttavia, molti studi hanno basato la loro scoperta di CIRCRNA sull’RNA totale e potrebbero quindi aver interpretato alcuni trascritti chimerici lineari come CIRCRNA, con conseguente rilevamento di circRNA falsi positivi. Per aggirare questo problema, la maggior parte degli studi ha confermato la circolarità dei trascritti trovati dall’RNA-seq totale utilizzando metodi RNasi R, Northern blot o elettroforetici (Jeck e Sharpless, 2014). Tuttavia, queste convalide di circolarità hanno anche talvolta rivelato trascrizioni che sembrano essere lineari, piuttosto che circolari confermando che la rilevazione di CIRCRNA a partire dall’RNA totale può portare ad alcuni falsi positivi. Questi falsi positivi sono stati attribuiti a artefatti tecnici o trascrizioni derivate da eventi non comuni come duplicazioni di esoni o eventi di trapianto (Jeck e Sharpless, 2014; Szabo e Salzman, 2016). Detto questo, l’opzione di aver trovato trascritti lineari veri, biologicamente attivi e funzionali che contengono una sequenza equivalente a una giunzione di backsplicing (da ora in poi chiamata trascritti lineari chimerici), è stata in qualche modo trascurata perché una fonte di tale RNA lineare non è stata conosciuta per le cellule sane.
I DNA circolari come fonte di trascritti lineari chimerici
La maggior parte del genoma umano è organizzata in cromosomi lineari, tuttavia alcune eccezioni sono state a lungo accettate come il DNA mitocondriale e le aberrazioni cromosomiche come i cerchi di DNA che trasportano oncogeni (ad esempio doppi minuti) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) e cromosomi ad anello (Tümer et al., 2004). Non è stato fino a poco tempo fa che diversi DNA circolari come MICRODNA (Shibata et al., 2012) o DNA circolare extracromosomiale (ECCDNA) sono stati trovati a derivare anche da grandi parti di diversi genomi eucariotici tra cui umani e lieviti (Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
I DNA circolari si formano quando due estremità di un DNA lineare sono unite insieme, dando luogo a una giunzione simile alla giunzione backspliced su circRNAs comunemente chiamata giunzione breakpoint che viene rilevata sulla base di varianti di lettura strutturale coerenti con un evento di circolarizzazione (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (Figura 1B). Di solito vanno da un centinaio di basi a cerchi megabase e possono contenere esoni e geni completi (Shibata et al ., 2012; Møller et al., 2015; Kumar et al., 2017; Turner et al., 2017; Møller et al., 2018) e mentre alcune regioni del genoma si trovano più comunemente sul DNA circolare (Sinclair e Guarente, 1997;; Møller et al., 2016; Turner et al., 2017; Møller et al., 2018), la maggior parte del DNA circolare sembra verificarsi a caso (Shibata et al., 2012; Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
È interessante notare che, in un recente articolo Møller et al. ha identificato migliaia di ECCDNA nei leucociti e nelle cellule muscolari nei controlli sani. Con l’idea di indagare se gli ECCDNA potessero essere trascritti, una libreria di mRNA è stata anche sequenziata dal tessuto muscolare e analizzata per gli eventi di trascrizione attraverso la giunzione del punto di interruzione dell’eccDNA rilevato trovando diverse corrispondenze (Møller et al., 2018). Questo risultato suggerisce che il DNA circolare nel tessuto sano viene trascritto, dando origine a trascritti lineari e poliadenilati che porteranno una sequenza equivalente alla sequenza di backsplicing dei CIRCRNA (Møller et al., 2018) (Figura 2).
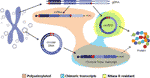
Figura 2 Rappresentazione grafica di diversi trascritti derivanti dal DNA genomico o dal DNA circolare. Gli esoni sono colorati in viola e le giunzioni backspliced o giunzioni chimeriche sono mostrati in rosso. I trascritti poliadenilati, chimerici contenenti giunzione e resistenti alla Rnasi R sono evidenziati rispettivamente in arancione, blu e giallo.
Le prove trascrizionali dei DNA circolari, insieme alla loro abbondanza, ci portano a suggerire che i DNA circolari potrebbero essere una fonte naturale di una notevole quantità di RNA lineari che trasportano giunzioni chimeriche. In molti casi, queste giunzioni chimeriche potrebbero essere indistinguibili dalle giunzioni backsplicing di CIRCRNA, e quindi potrebbero essere fattori confondenti negli studi circRNA. Nei paragrafi seguenti, spiegheremo i dati a sostegno di questa proposta.
circRNA di rilevamento: Tutto ciò che luccica non è oro
Come precedentemente introdotto, i CIRCRNA si formano attraverso un evento di splicing non canonico chiamato backsplicing. Le trascrizioni risultanti da questo evento di backsplicing hanno una struttura ad anello covalentemente chiusa senza polarità 5′-3′, né una coda poliadenilata e, cosa più importante, sono caratterizzate dalla presenza di un ordine di esoni criptati rispetto alla trascrizione lineare (Zhang et al., 2016; Wilusz, 2018). Questo ordine di esone strapazzate diventa evidente nella giunzione backspliced che collega un 5 ‘sequenza a valle con un upstream 3’ sequenza. Pertanto, tutti gli algoritmi di rilevamento circRNA sfruttano la presenza delle giunzioni back-spliced come caratteristica diagnostica per l’identificazione di circRNA (Figura 1A).
Diversi metodi sono stati adattati per il rilevamento di queste giunzioni back-spliced. Gli array commerciali contenenti sonde che mirano a queste regioni backspliced sono stati ampiamente utilizzati negli studi di screening dei biomarcatori (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). La successiva validazione si basa spesso anche sull’amplificazione delle giunzioni di backsplicing utilizzando primer divergenti (Panda e Gorospe, 2018). Molti altri documenti hanno condotto un’analisi di sequenziamento ad alto throughput che supera uno dei principali limiti degli array consentendo di rilevare non solo i CIRCRNA annotati ma anche gli eventi di circolarizzazione dell’RNA de novo da regioni genomiche in cui nessun circRNA è stato annotato da studi precedenti. Diverse condutture bioinformatiche sono state sviluppate per la rilevazione di CIRCRNA nei set di dati RNA-Seq, ma tutte sono basate sulla presenza di letture che attraversano le giunzioni di back-splicing e trovare quella più affidabile è ancora una sfida per i bioinformatici (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
È possibile seguire due approcci principali per la rilevazione di CIRCRNA nei dati RNA-Seq. In primo luogo, molti studi circRNA RNA-seq si basano su campioni trattati con RNasi R al fine di esaurire tutti gli RNA lineari prima del sequenziamento. Sebbene questo approccio sia appositamente progettato per il rilevamento del circRNA, vale la pena notare che la degradazione della RNasi R è variabile, che ci sono rari casi di RNA lineari resistenti alla RNasi R e CIRCRNA sensibili alla RNasi R (Szabo e Salzman, 2016) e che questo trattamento richiede un elevato input di RNA che potrebbe essere limitante per alcuni tessuti. Altri studi su circRNA scelgono di sequenziare l’RNA totale, ribosomiale impoverito (ribo -) o non poliadenilato (polyA -), dove si possono trovare sia RNA lineari che circolari (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak et al., 2015). Questo approccio evita l’uso di RNasi R, che riduce la quantità di RNA necessaria per il sequenziamento e consente di studiare l’espressione di altri tipi di RNA dallo stesso set di dati. È stato dimostrato che con una buona profondità e qualità di sequenziamento e un’attenta analisi dei dati, i veri CIRCRNA possono essere rilevati dal sequenziamento totale dell’RNA (Wang et al., 2017), tuttavia, in questo secondo approccio, è necessaria una successiva conferma di circolarità per i CIRCRNA.
Con la scoperta di RNA chimerici lineari trascritti da DNA circolari, i CIRRNA non sono più gli unici trascritti con giunzioni chimeriche. Pertanto, è della massima importanza notare che mentre il primo approccio arricchirà significativamente il campione di RNA nei CIRCRNA in modo che la maggior parte delle giunzioni chimeriche rilevate corrispondano ai veri CIRCRNA, il secondo potrebbe sovrastimare il numero di trascritti circRNA attribuendo ai CIRCRNA il segnale proveniente sia dai CIRCRNA che dai trascritti chimerici lineari trascritti dai DNA circolari. Di conseguenza, tenendo conto della coesistenza di CIRCRNA e trascrizioni chimeriche lineari, la necessità di test di circolarità e test di funzionalità acquista importanza e si dovrebbe prestare particolare attenzione non solo ai metodi sperimentali ma anche computazionali per evitare di confondere trascrizioni chimeriche da DNA circolari con CIRCRNA formate da backsplicing.
Discussione
Il campo circRNA è ancora in una fase iniziale, tuttavia, i CIRCRNA hanno già dimostrato di essere molecole sorprendenti, implicate in molti processi, con un grande potenziale di biomarcatore e che possono anche cambiare il modo in cui comprendiamo i processi di trascrizione e traduzione. Per questi motivi, stanno guadagnando attenzione e il campo circRNA è al momento uno dei campi di ricerca sull’RNA più attivi. Tuttavia, ci sono ancora molti conflitti, controversie e domande aperte (Li, 2019) che devono essere discusse.
In questo rapporto, e alla luce dei recenti progressi nel campo del DNA circolare, vogliamo sottolineare la trascrizione dal DNA circolare extracromosomiale come una delle principali fonti naturali di trascritti lineari con segnali back-spliced che potrebbero interferire con i dati circRNA (Møller et al., 2018). D’ora in poi, a parte gli artefatti tecnici, le duplicazioni e gli eventi di trapianto che potrebbero portare a falsi positivi nel rilevamento del circRNA, dovremmo tenere conto anche dell’esistenza di questo nuovo tipo di trascrizioni chimeriche. Pertanto i test di circolarizzazione e i test funzionali sono più importanti che mai.
In ogni caso, questi trascritti lineari chimerici non dovrebbero essere considerati solo come un semplice fattore di confusione per gli studi circRNA. Nonostante le implicazioni tecniche per la caratterizzazione del circRNA, l’esistenza di queste molecole di RNA lineari chimeriche simili a circRNA provenienti da ECCDNA aggiunge un nuovo tipo di molecola alla lista sempre crescente di RNA e amplia la nostra visione sulla complessità del trascrittoma e la sua regolazione. Inoltre, queste molecole di RNA lineari provenienti da eccDNA potrebbero anche presentare funzioni simili al circRNA, comprese le funzioni regolatorie o il potenziale da tradurre. I prodotti genici dei trascritti eccDNA potrebbero potenzialmente contribuire al fenotipo delle cellule somatiche e dei tessuti come riportato nel lievito (Gresham et al., 2010; Demeke et al., 2015). Tuttavia, in questo campo nascente, sono necessari più dati e ricerche per iniziare a graffiare la superficie dell’iceberg.
Contributi dell’autore
LI, IP-L, BR e DO hanno scritto il documento.
Finanziamento
Questo studio è stato finanziato dall’Instituto de Salud Carlos III attraverso il progetto “PI17/00189” (cofinanziato dal Fondo Europeo di Sviluppo Regionale/Fondo Sociale Europeo) “Investire nel tuo futuro”). IP – L e BR sono stati sostenuti dal Consiglio danese per la ricerca indipendente, 6108-00171B e LI è stato sostenuto dal Dipartimento dell’Educazione del governo basco .
Conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di rapporti commerciali o finanziari che potrebbero essere interpretati come un potenziale conflitto di interessi.