significantie
er is een groeiende belangstelling voor circRNAs vanwege hun implicatie in vele biologische processen en ziekten naast hun biomarker potentieel. Ze worden voornamelijk gedetecteerd door de aanwezigheid van Leest mapping hun backsplicing junction. Niettemin, zijn circRNAs niet langer de enige afschriften die dergelijke verbinding bevatten aangezien de recente studies hebben geopenbaard dat cirkeldna ‘ s gemeenschappelijk zijn en kunnen worden getranscribeerd resulterend in afschriften die een circRNA-signaal zouden nabootsen. Daarom kan dit nieuwe type chimerische transcript de manier waarop circRNA analyse wordt gedaan veranderen en invloed hebben op een aantal van de reeds gerapporteerde resultaten.
zijn circulaire Rnals de enige Chimerische transcripten?
circulair RNA (circRNAs) werden enkele jaren geleden herontdekt als niet-canonisch gesplitste RNA-vormen die aanwezig zijn in verschillende organismen, waaronder mensen (Salzman et al., 2012; Jeck et al., 2013). Zij zijn covalent gesloten afschriften die door een achter-verbindende gebeurtenis van RNA worden gevormd, waar een lasdonor van stroomafwaartse exon aan een stroomopwaartse lasacceptor aansluit leidend tot covalent gesloten afschriften die door de aanwezigheid van een achter-verbindende verbinding worden gekenmerkt die circRNAs van hun lineaire tegenhangers (figuur 1A) (Zhang et al., 2016; Wilusz, 2018).
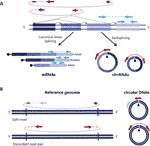
figuur 1 De opsporing van zowel cirkelrna als DNAs is gebaseerd op leest die de verbinding overspannen. (A) Backspliced verbinden tussen exons en canonical exon verbinden worden afgebeeld met niet-continue en continue lijnen respectievelijk aanleiding geven tot circRNAs en mRNAs. Paired-end reads verspreid over de backspliced junctions worden weergegeven in rood en paired-end reads consistent met een lineaire transcript worden weergegeven in blauw. (B) gelijkaardig aan circRNAs, worden cirkeldna ‘ s (eccDNAs) ontdekt gebaseerd op structureel-gelezen varianten consistent met een circularisatiegebeurtenis die in rood wordt afgebeeld .
sinds hun herontdekking, heeft de wetenschappelijke gemeenschap haar aandacht gevestigd op circRNAs en heeft hun betrokkenheid bij verschillende cellulaire processen in gezondheid en ziekte onderzocht (Haque en Harries, 2017), hun potentiële rol als biomarkers (Abu en Jamal, 2016), en hun regulerende functies (Floris et al., 2016). CircRNAs zijn nu bekend overvloedig en stabiel in de cytosol en de kern (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) en zijn ook vrij gevonden in biofluids (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) en in extracellulaire blaasjes (Kyoung Mi et al., 2017). Het biomarkerpotentieel van circRNAs is intens bestudeerd, in feite, zijn er gepubliceerd vele case-controlestudies die voor differentieel uitgedrukt circRNAs zoeken die biomarkers van verschillende ziekten zouden kunnen zijn. Tot op heden zijn circRNAs betrokken bij verschillende ziekten, waaronder kanker (Kristensen et al., 2017; Arnaiz et al., 2018), neurologische aandoeningen (Akhter, 2018), hart-en vaatziekten (Aufiero et al., 2019) en immuungerelateerde ziekten (Iparraguirre et al., 2017; Liu et al., 2019). Tegelijkertijd blijven hun biogenese, Kenmerken, Functies en implicaties in de menselijke biologie volledig begrijpen als open vragen voor onderzoekers op het gebied.
hoewel de functie van de meeste circRNAs onbekend blijft, is aangetoond dat sommige circRNAs kunnen fungeren als microRNA sponzen, die de microRNA niveaus en hun activiteit reguleren (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Ze zijn betrokken bij genexpressie Regulatie door het reguleren van de transcriptie van hun ouderlijke genen, concurreren met de lineaire splicing of sponzing eiwitten (Ashwal-Fluss et al., 2014; Li et al., 2018). Interessant, ribosoom profileren studies hebben onlangs aangetoond dat circRNAs ook kan worden vertaald zowel in vitro als in vivo (Legnini et al., 2017; Yang et al., 2017).
het belangrijkste kenmerk van circRNAs, en verantwoordelijk voor de meeste van hun speciale eigenschappen, is hun circulariteit. Daarom, naast het ontdekken van hun karakteristieke achter-gesplitste verbinding, het testen van de circulariteit van deze moleculen, is een van de belangrijkste punten in elke circRNA-studie. Niettemin, hebben vele studies hun ontdekking van circRNAs op totaal RNA gebaseerd en zouden daardoor sommige lineaire chimeric transcripten als circRNAs kunnen hebben geïnterpreteerd, resulterend in vals-positieve circRNA-detecties. Om dit probleem te omzeilen hebben de meeste studies de circulariteit van de afschriften bevestigd die door totaal RNA-seq worden gevonden gebruikend RNase R, Noordelijke vlek of elektroforetische methodes (Jeck en Sharpless, 2014). Nochtans, hebben deze circulariteitsvalidaties ook soms transcripties geopenbaard die lineair lijken te zijn, eerder dan cirkelvormig bevestigend dat de opsporing van circRNAs beginnend van totaal RNA tot sommige valse positieven kan leiden. Deze valse positieven zijn toegeschreven aan technische artefacten of transcripties afgeleid van ongewone gebeurtenissen zoals exon duplicaties of transplicing gebeurtenissen (Jeck and Sharpless, 2014; Szabo and Salzman, 2016). Dat gezegd hebbende, is de optie om ware, biologisch actieve en functionele lineaire transcripten te hebben gevonden die een opeenvolging gelijkwaardig aan een backsplicing junction (van nu af aan genoemd chimeric lineaire transcripten) bevatten, enigszins over het hoofd gezien omdat een bron van dergelijk lineair RNA niet voor gezonde cellen gekend is.
circulaire DNA ‘ s als bron van Chimerische Lineaire transcripten
het grootste deel van het menselijk genoom is georganiseerd in lineaire chromosomen, echter, enkele uitzonderingen zijn al lang geaccepteerd, zoals mitochondriaal DNA, en chromosomale afwijkingen zoals DNA-cirkels die oncogenen dragen (bijvoorbeeld dubbele minuten) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) en ringchromosomen (Tümer et al., 2004). Het was niet tot voor kort dat verschillende circulaire DNA ‘ s zoals microDNAs (Shibata et al., 2012) of extrachromosomale circulaire DNAs (eccDNAs) werden gevonden om ook voortvloeien uit grote delen van verschillende eukaryotic genomen met inbegrip van menselijke en gist (Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
cirkelvormige DNA ‘ s worden gevormd wanneer twee uiteinden van een lineair DNA gezamenlijk worden gevormd, wat resulteert in een kruising die vergelijkbaar is met de achtersplitste kruising op circRNAs die gewoonlijk breekpuntknooppunt wordt genoemd en die wordt gedetecteerd op basis van structureel-gelezen varianten die consistent zijn met een circularisatiegebeurtenis (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (figuur 1B). Ze variëren meestal van honderd basen tot megabase cirkels en kunnen volledige exonen en genen bevatten (Shibata et al ., 2012; Møller et al., 2015; Kumar et al., 2017; Turner et al., 2017; Møller et al., 2018) en terwijl sommige gebieden van het genoom vaker worden gevonden op circulaire DNA (Sinclair and Guarente, 1997;; Møller et al., 2016; Turner et al., 2017; Møller et al., 2018), lijken de meeste cirkelvormige DNA willekeurig voor te komen (Shibata et al., 2012; Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
interessant, in een recente paper Møller et al. identificeerde duizenden eccDNAs in leukocyten en spiercellen in gezonde controles. Met het idee om te onderzoeken of eccDNAs kunnen worden getranscribeerd, werd een mRNA bibliotheek ook gesequenced van spierweefsel en geanalyseerd voor transcriptie gebeurtenissen over de breekpunt kruising van de gedetecteerde eccDNA vinden van verschillende wedstrijden (Møller et al., 2018). Deze bevinding stelt voor dat circulaire DNA in gezond weefsel wordt getranscribeerd, die leiden tot lineaire en polyadenylated transcripten die een opeenvolging gelijkwaardig aan de backsplicing opeenvolging van circRNAs (Møller et al., 2018) (Figuur 2).
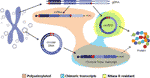
Figuur 2 grafische weergave van verschillende afschriften arised van of genomic DNA of cirkeldna. Exons zijn gekleurd in paars en de backspliced junctions of chimeric junctions zijn weergegeven in rood. Polyadenylated, chimeric verbinding die en RNase R bestand afschriften bevatten worden gemarkeerd in oranje, blauw en geel respectievelijk.
het transcriptionele bewijs van circulaire DNA ‘s, samen met hun overvloed, leidt ons om te suggereren dat circulaire DNA’ s een natuurlijke bron zouden kunnen zijn van een aanzienlijke hoeveelheid lineaire RNA ‘ s die chimerische kruispunten dragen. In veel gevallen kunnen deze chimerische juncties niet te onderscheiden zijn van de backsplicing junctions van circRNAs, en daarom kunnen ze verstorende factoren zijn in circRNA-studies. In de volgende paragrafen zullen we de gegevens die dit voorstel ondersteunen toelichten.
circRNA-detectie: Alles wat glittert Is niet Goud
zoals eerder geà ntroduceerd, worden circRNAs gevormd door een niet-canonieke splicing event genaamd backsplicing. Transcripten als gevolg van deze backsplicing gebeurtenis hebben een covalent gesloten lus structuur met noch 5′ -3 ‘ polariteit, noch een polyadenylated staart en nog belangrijker, ze worden gekenmerkt door de aanwezigheid van een versleutelde exon orde ten opzichte van de lineaire transcript (Zhang et al., 2016; Wilusz, 2018). Deze scrambled exon volgorde wordt duidelijk in de backspliced junction die een 5’ downstream sequentie verbindt met een upstream 3 ‘ sequentie. Aldus, exploiteren alle circRNA-opsporingsalgoritmen de aanwezigheid van de achter-gesplitste verbindingen als kenmerkende eigenschap voor de identificatie van circRNA (figuur 1A).
verschillende methoden zijn aangepast voor de detectie van deze back-spliced junctions. De commerciële reeksen die sondes bevatten die deze achtersplitste gebieden richten zijn wijd gebruikt in biomarkeronderzoek studies (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). De daaropvolgende validatie is vaak ook gebaseerd op de versterking van de backsplicing junctions met behulp van divergente primers (Panda en Gorospe, 2018). Vele andere documenten hebben een hoge productie geleid die analyse rangschikken die één van de belangrijkste beperkingen van de reeksen overwint die niet alleen de geannoteerde circRNAs maar ook de circularisatiegebeurtenissen van novo RNA van genomic gebieden toestaan waar geen circRNA door Vorige studies werd geannoteerd. Verscheidene bioinformatische pijpleidingen zijn ontwikkeld voor de opsporing van circRNAs in datasets van RNA-Seq, maar allen zijn gebaseerd op de aanwezigheid van Leest die over de rug-verbindende verbindingen kruisen en het vinden van betrouwbaarste nog een uitdaging voor bioinformaticians (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
twee belangrijke benaderingen kunnen voor de opsporing van circRNAs in RNA-Seq gegevens worden gevolgd. Ten eerste, zijn vele circRNA RNA-seq studies gebaseerd op RNase R behandelde steekproeven om alle lineaire RNAs te deplete alvorens te rangschikken. Hoewel deze benadering speciaal voor de circRNA-opsporing wordt ontworpen is het vermeldenswaard dat de degradatie van RNase R variabel is, dat er zeldzame gevallen van RNase R bestand lineaire RNAs en RNase R gevoelige circRNAs (Szabo en Salzman, 2016) zijn en dat deze behandeling een hoge input van RNA vereist die voor sommige weefsels zou kunnen beperken. Andere circRNA-studies kiezen ervoor om ofwel totaal, ribosomaal-uitgeput (ribo-), of niet-polyadenylated (polyA-) RNA te sequencen, waar zowel lineaire als cirkelvormige RNAs kunnen worden gevonden (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak et al., 2015). Deze benadering vermijdt het gebruik van RNase R, die het bedrag van RNA vermindert nodig voor het rangschikken en staat toe het bestuderen van de uitdrukking van andere types van RNAs van dezelfde dataset toe. Men heeft aangetoond dat met het goede rangschikken diepte en kwaliteit en een zorgvuldig gegevens-analyse, ware circRNAs van totaal RNA kan worden ontdekt rangschikkend (Wang et al., 2017), echter, in deze tweede benadering, een latere circulariteit bevestiging voor circRNAs nodig is.
met de ontdekking van lineaire Chimerische RNAs getranscribeerd uit circulaire DNA ‘ s, circRNAs zijn niet langer de enige transcripten met chimerische juncties. Daarom is het van het grootste belang om op te merken dat terwijl de eerste benadering de steekproef van RNA in circRNAs beduidend zal verrijken zodat de meeste gedetecteerde chimerische verbindingen met ware circRNAs zullen corresponderen, kan de tweede het aantal circRNA-transcripten overschatten door aan circRNAs het signaal toe te schrijven dat van zowel circRNAs komt als de lineaire chimerische transcripten die van circrnas worden getranscribeerd. Bijgevolg, rekening houdend met de coëxistentie van circRNAs en lineaire chimerische transcripties, de behoefte aan circulariteitstests en functionaliteitsanalyses winsten belang en speciale zorg moet worden genomen met betrekking tot niet alleen experimentele maar ook computationele methoden om te vermijden verwarren chimerische transcripties van circulaire DNAs met circRNAs gevormd door backsplicing.
discussie
het circRNA-veld bevindt zich nog in een vroeg stadium, maar circRNAs heeft al aangetoond verbazingwekkende moleculen te zijn, betrokken bij vele processen, met een groot biomarkerpotentieel en dat kan ook de manier waarop we de transcriptie-en vertaalprocessen begrijpen veranderen. Om deze redenen, winnen zij aandacht en het circRNA-gebied is op het ogenblik één van de meest actieve onderzoeksgebieden van RNA. Er zijn echter nog veel conflicten, controverses en open vragen (Li, 2019) die besproken moeten worden.
in dit rapport, en in het licht van de recente ontwikkelingen in het circulaire DNA-veld, willen we wijzen op de transcriptie van extrachromosomaal circulaire DNA als een van de belangrijkste natuurlijke bronnen van lineaire transcripten met back-gesplitste signalen die kunnen interfereren met circRNA data (Møller et al., 2018). Van nu af aan, afgezien van de technische artefacten, duplicaties en transplicing gebeurtenissen die kunnen leiden tot valse positieven in de circRNA detectie, moeten we ook rekening houden met het bestaan van dit nieuwe type van chimerische transcripten. Daarom circularisatietests en functionele analyses zijn belangrijker dan ooit.
in ieder geval moeten deze chimerische lineaire transcripties niet alleen worden beschouwd als een louter verstorende factor voor circRNA-studies. Ondanks de technische implicaties voor de circRNA karakterisatie, voegt het bestaan van deze circRNA-achtige chimerische lineaire RNA moleculen afkomstig van eccDNAs een nieuw type molecule toe aan de steeds groeiende lijst van RNAs en breidt onze visie over de complexiteit van het transcriptoom en zijn Regulatie uit. Bovendien konden deze lineaire molecules van RNA die van eccDNA komen ook functies voorstellen gelijkend op circRNA, met inbegrip van de regelgevende functies of het te vertalen potentieel. Genproducten uit eccdna transcripten kunnen mogelijk bijdragen aan het fenotype van somatische cellen en weefsel zoals gerapporteerd in gist (Gresham et al., 2010; Demeke et al., 2015). In dit ontluikende veld is echter meer data en onderzoek nodig om de ijsberg te kunnen krassen.
Auteursbijdragen
LI, IP-L, BR En DO schreven de paper.
financiering
deze studie werd gefinancierd door het Instituto De Salud Carlos III via het project “PI17/00189” (medegefinancierd door het Europees Fonds voor Regionale Ontwikkeling/Europees Sociaal Fonds) “investeren in uw toekomst”). IP-L en BR werden gesteund door de Deense Raad voor onafhankelijk onderzoek, 6108-00171B en LI werd gesteund door het Ministerie van Onderwijs van de Baskische regering .
belangenconflict
de auteurs verklaren dat het onderzoek werd uitgevoerd zonder enige commerciële of financiële relatie die als een potentieel belangenconflict kon worden opgevat.