Lunes, 21 de septiembre de 2020
Incluso en el campo de la genómica, donde se producen nuevos avances cada pocos meses, la finalización del primer autosoma humano completamente secuenciado es un logro trascendental. Altamente preciso, sin huecos, sin uniones erróneas, solo el cromosoma 8 en todo su esplendor. Es una hazaña notable y nos sentimos honrados de que PacBio HiFi reads haya desempeñado un papel fundamental para ayudar a lograrlo.
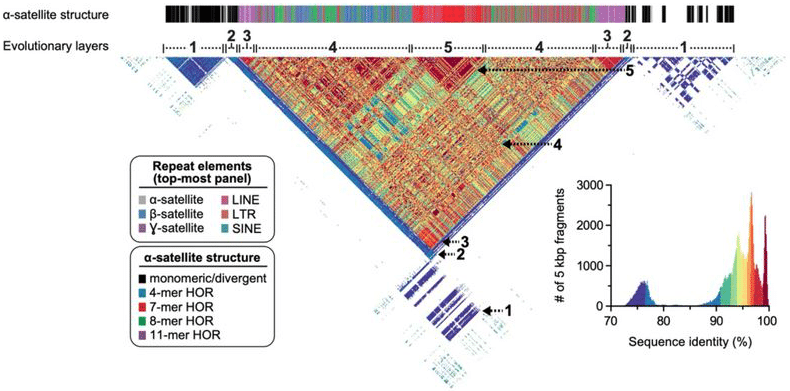
La secuencia completa de centrómeros del cromosoma 8 muestra una diversidad de repeticiones satelitales y otras repeticiones genómicas abundantes, ahora con una resolución de nivel de base casi perfecta de extremo a extremo. Logsdon, G et al. (2020)
Este trabajo se describe en un preprint publicado recientemente en bioRxiv por el autor principal Glennis Logsdon (@glennis_logsdon), el autor principal Evan Eichler y sus colaboradores en el Consorcio Telómero a Telómero (T2T). Es parte de la iniciativa T2T más amplia para secuenciar y ensamblar el primer genoma humano verdaderamente completo y sigue la liberación anterior del cromosoma X completamente secuenciado.
«Desde el anuncio de la secuenciación del genoma humano hace 20 años, los cromosomas humanos han permanecido inacabados debido a grandes regiones de repeticiones altamente idénticas ubicadas dentro de centrómeros, duplicación segmentaria y los brazos cortos acrocéntricos de los cromosomas», señalan los autores. «El advenimiento de tecnologías de secuenciación de larga lectura y algoritmos asociados ahora han hecho posible ensamblar sistemáticamente estas regiones a partir de ADN nativo por primera vez.»
El cromosoma 8 hizo un objetivo atractivo para el primer autosoma del T2T debido a su centrómero manejable (previamente estimado en 1,5 Mb a 2,2 Mb de largo). Pero el cromosoma es también el hogar de » una de las regiones estructuralmente más dinámicas del genoma humano—el cúmulo de genes β-defensina ubicado en 8p23.1—así como un neocentrómero ubicado en 8q21.2, que han estado en gran parte sin resolver durante los últimos 20 años», escriben los científicos. El grupo de β-defensin juega un papel clave en la inmunidad innata y la variación estructural en esta región ha estado implicada durante mucho tiempo en la enfermedad humana.
El nuevo ensamblaje, que aborda los cinco vacíos previamente intratables en el genoma humano de referencia, se construyó con un método inteligente que utiliza varios conjuntos de datos, incluidas lecturas largas precisas: «Más de la mitad de los datos de PacBio HiFi están contenidos en lecturas superiores a 17,8 kbp, con una precisión media superior al 99,9%.»Después de un escalón de andamiaje basado en lecturas de nanoporos de Oxford, se intercambiaron contiguos ensamblados a partir de lecturas de alta fidelidad de PacBio para proporcionar la resolución de par de bases. «Mejoramos la precisión del par de bases de los andamios de secuencia reemplazando la secuencia ONT sin procesar con varios contigs concordantes de PacBio HiFi», informa el equipo.
La secuencia completa de chr8 se ajusta a 146 Mb e incluye más de 3 Mb que faltan en GRCh38. Como Logsdon et al. escribe: «El resultado es un ensamblaje de cromosomas completos con una precisión estimada de pares de bases superior al 99,99%.»
Los científicos también abordaron ese cúmulo de genes de β-defensina, «que resolvimos en un único locus de 7,06 Mbp, sustancialmente mayor que la región de 4,56 Mbp en el genoma humano de referencia actual», señalan. Casi todos esos datos de secuencia, el 99,9934%, para ser precisos, provenían de lecturas de alta fidelidad. El centrómero completo, por su parte, representaba 2,08 Mb.
Con este hermoso conjunto en la mano, el equipo de T2T lo sacó a dar una vuelta. Primero, lo validaron con una serie de herramientas ortogonales, como el mapeo óptico. A continuación, generaron datos de alta fidelidad para los ortólogos del cromosoma 8 en chimpancés, macacos y orangutanes para comparar los datos de la secuencia y reconstruir la historia evolutiva del autosoma humano. «Los análisis comparativos y filogenéticos muestran que la estructura de satélite α de orden superior evolucionó específicamente en el ancestro de los grandes simios, y la región centromérica evolucionó con una simetría en capas», escribe el equipo. «Estimamos que la tasa de mutación del ADN satelital centromérico se acelera al menos 2.2 veces, y esta aceleración se extiende más allá del satélite α de orden superior hacia la secuencia de flanqueo.»
Finalmente, los investigadores realizaron un análisis de transcripciones completas producidas con el método Iso-Seq. Ese proceso identificó «61 loci codificadores de proteínas y 33 loci no codificadores que se corresponden mejor con esta secuencia de cromosoma 8 terminada que con GRCh38, incluido el descubrimiento de nuevos genes que se mapean para copiar regiones polimórficas numéricas», informan. Doce de estos nuevos genes fueron descubiertos solo en ese delicado locus β-defensin.
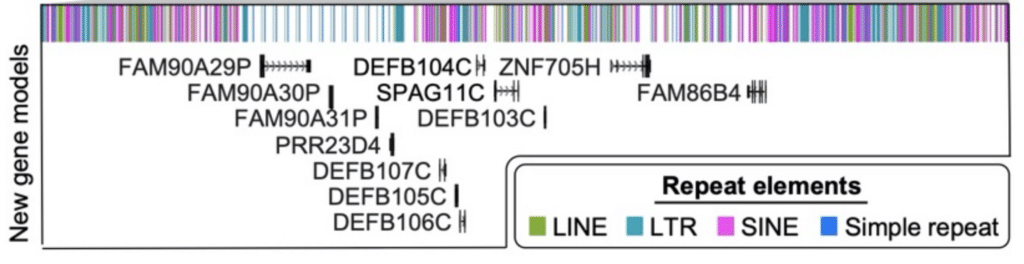
Una combinación de ensamblaje del genoma HiFi y anotación de ARN con datos Iso-Seq agregó estos 12 nuevos genes a la región β-defensin (DEFB) del cromosoma 8. Logsdon, G et al. (2020)
Para muchos de nosotros en la comunidad de la genómica, este documento representa mucho más que la secuencia de un solo cromosoma humano. Es una declaración sobre lo que la ciencia puede lograr ahora y hacia dónde nos puede llevar en los años venideros. Como resumieron los autores: «Ahora que regiones complejas como estas se pueden secuenciar y ensamblar, será importante extender estos análisis a otros centrómeros, múltiples individuos y especies adicionales para comprender su impacto completo con respecto a la variación y evolución genética.»
Puede escuchar más detalles de Logsdon directamente en una conferencia en línea gratuita organizada conjuntamente por el Consorcio T2T y el Consorcio de Referencia de Pangenomas Humanos (HPRC) el 22 y 23 de septiembre. Los oradores ofrecerán nuevas ideas sobre el cromosoma 8 e informarán sobre el progreso de la T2T hacia un ensamblaje completo del genoma humano. En el mismo evento, el HPRC presentará su esfuerzo complementario para secuenciar cientos de genomas humanos de alta calidad. Los presentadores incluyen: Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) y muchos más. Estaremos pegados a nuestros ordenadores para ello y esperamos que también tengas la oportunidad de unirte.