Significación
Existe un creciente interés en los circRNAs debido a su implicación en muchos procesos biológicos y enfermedades, además de su potencial biomarcador. Se detectan principalmente por la presencia de lecturas que mapean su unión de corte posterior. Sin embargo, los ARN circulares ya no son las únicas transcripciones que contienen tal unión, ya que estudios recientes han revelado que los ARN circulares son comunes y pueden ser transcritos, lo que resulta en transcripciones que imitan una señal de ARN circular. Por lo tanto, este nuevo tipo de transcripción quimérica puede cambiar la forma en que se está haciendo el análisis del circRNA e impactar algunos de los resultados ya reportados.
¿Son los ARN Circulares las Únicas Transcripciones Quiméricas?
El ARN circular (circRNAs) se redescubrió hace unos años como formas de ARN no empalmadas canónicamente presentes en diferentes organismos, incluidos los humanos (Salzman et al., 2012; Jeck et al., 2013). Son transcripciones covalentemente cerradas formadas a través de un evento de empalme posterior de ARN, donde un donante de empalme de un exón aguas abajo se une a un aceptor de empalme aguas arriba que conduce a transcripciones covalentemente cerradas que se caracterizan por la presencia de una unión de empalme posterior que hace que los circRNAs se distingan de sus contrapartes lineales (Figura 1A) (Zhang et al., 2016; Wilusz, 2018).
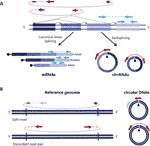
Figura 1 La detección de ARN circular y ADN se basa en lecturas que abarcan la unión. (A) La unión a contracorriente entre exones y la unión canónica de exones se representa con líneas no continuas y continuas, respectivamente, dando lugar a circRNAs y mRNAs. Las lecturas de los extremos emparejados que abarcan las uniones de la pared posterior se muestran en rojo y las lecturas de los extremos emparejados consistentes con una transcripción lineal se muestran en azul. B) De manera similar a los circRNAs, se detectan ADN circulares (ECDNAS) sobre la base de variantes de lectura estructural consistentes con un evento de circularización representado en rojo .
Desde su redescubrimiento, la comunidad científica ha llamado su atención sobre los circRNAs y ha investigado su participación en varios procesos celulares en la salud y la enfermedad (Haque y Harries, 2017), su papel potencial como biomarcadores (Abu y Jamal, 2016) y sus funciones reguladoras (Floris et al., 2016). Ahora se sabe que los circRNAs son abundantes y estables en el citosol y el núcleo (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) y también se han encontrado libres en biofluidos (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) y en vesículas extracelulares (Kyoung Mi et al., 2017). El potencial de biomarcadores de los circRNAs ha sido intensamente estudiado, de hecho, se han publicado muchos estudios de casos y controles que buscan circRNAs expresados diferentemente que podrían ser biomarcadores de diferentes enfermedades. Hasta la fecha, los circRNAs han estado implicados en varias enfermedades, incluido el cáncer (Kristensen et al., 2017; Arnaiz et al., 2018), trastornos neurológicos (Akhter, 2018), enfermedades cardiovasculares (Aufiero et al., 2019) y enfermedades relacionadas con el sistema inmunitario (Iparraguirre et al., 2017; Liu et al., 2019). Al mismo tiempo, llegar a comprender completamente su biogénesis, características, funciones e implicaciones en la biología humana sigue siendo una pregunta abierta para los investigadores en el campo.
Aunque la función de la mayoría de los circRNAs sigue siendo desconocida, se ha demostrado que algunos circRNAs pueden actuar como esponjas de microRNA, regulando los niveles de microRNA y su actividad (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Participan en la regulación de la expresión génica regulando la transcripción de sus genes parentales, compitiendo con las proteínas de empalme lineal o esponjado (Ashwal-Fluss et al., 2014; Li et al., 2018). Curiosamente, los estudios de perfiles de ribosomas han demostrado recientemente que los circRNAs también se pueden traducir tanto in vitro como in vivo (Legnini et al., 2017; Yang et al., 2017).
La característica principal de los circRNAs, y responsable de la mayoría de sus propiedades especiales, es su circularidad. Por lo tanto, además de detectar su unión de empalme posterior característica, probar la circularidad de estas moléculas es uno de los puntos clave en cada estudio de circRNA. Sin embargo, muchos estudios han basado su descubrimiento de circRNAs en ARN total y, por lo tanto, podrían haber interpretado algunas transcripciones quiméricas lineales como circRNAs, lo que resulta en detecciones de circRNAs falsas positivas. Para evitar este problema, la mayoría de los estudios han confirmado la circularidad de las transcripciones encontradas por ARN-seq total utilizando RNasa R, Northern blot o métodos electroforéticos (Jeck y Sharpless, 2014). Sin embargo, estas validaciones de circularidad también han revelado a veces transcripciones que parecen ser lineales, en lugar de circulares, confirmando que la detección de ARN a partir del ARN total puede conducir a algunos falsos positivos. Estos falsos positivos se han atribuido a artefactos técnicos o transcripciones derivadas de eventos poco comunes, como duplicaciones de exones o eventos de trasplante (Jeck y Sharpless, 2014; Szabo y Salzman, 2016). Dicho esto, la opción de haber encontrado transcripciones lineales verdaderas, biológicamente activas y funcionales que contienen una secuencia equivalente a una unión de repetición (de ahora en adelante llamada transcripciones lineales quiméricas), ha sido algo pasada por alto porque una fuente de tal ARN lineal no se ha conocido para las células sanas.
ADN circular como Fuente de Transcripciones Lineales Quiméricas
La mayor parte del genoma humano está organizado en cromosomas lineales, sin embargo, algunas excepciones han sido aceptadas durante mucho tiempo, como el ADN mitocondrial, y aberraciones cromosómicas como los círculos de ADN que llevan oncogenes (por ejemplo, minutos dobles) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) y cromosomas de anillo (Tümer et al., 2004). No fue hasta hace poco que diferentes ADN circulares como microDNAs(Shibata et al., 2012) o ADN circular extracromosómico (eccDNAs) también surgieron de grandes partes de diferentes genomas eucarióticos, incluidos humanos y levaduras (Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
Los ADN circulares se forman cuando dos extremos de un ADN lineal se unen, lo que resulta en una unión similar a la unión de la pared posterior en los circRNAs, comúnmente llamada unión de punto de ruptura, que se detecta en función de variantes de lectura estructural consistentes con un evento de circularización (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (Figura 1B). Por lo general, van desde cien bases hasta círculos de megabase y pueden contener exones y genes completos (Shibata et al ., 2012; Møller et al., 2015; Kumar et al., 2017; Turner et al., 2017; Møller et al., 2018)y aunque algunas regiones del genoma se encuentran más comúnmente en el ADN circular (Sinclair y Guarente, 1997;; Møller et al., 2016; Turner et al., 2017; Møller et al., 2018), la mayoría del ADN circular parece ocurrir al azar (Shibata et al., 2012; Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
Curiosamente, en un artículo reciente Møller et al. se identificaron miles de ECDNAS en leucocitos y células musculares en controles sanos. Con la idea de investigar si los ECDNAS podían ser transcritos, también se secuenció una biblioteca de ARNm a partir de tejido muscular y se analizó para detectar eventos de transcripción a través de la unión del punto de ruptura del ECDNA detectado, encontrando varias coincidencias (Møller et al., 2018). Este hallazgo sugiere que el ADN circular en tejido sano se transcribe, dando lugar a transcripciones lineales y poliadeniladas que llevarán una secuencia equivalente a la secuencia de corte posterior de los circRNAs (Møller et al., 2018) (Figura 2).
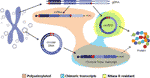
Figura 2 Representación gráfica de diferentes transcripciones obtenidas a partir de ADN genómico o ADN circular. Los exones son de color morado y las uniones con lámina posterior o uniones quiméricas se muestran en rojo. Las transcripciones poliadeniladas que contienen uniones quiméricas y resistentes a la RNasa R se resaltan en naranja, azul y amarillo, respectivamente.
La evidencia transcripcional de los ADN circulares, junto con su abundancia, nos lleva a sugerir que los ADN circulares podrían ser una fuente natural de una cantidad sustancial de ARN lineales que llevan uniones quiméricas. En muchos casos, estas uniones quiméricas podrían ser indistinguibles de las uniones de corte posterior de los circRNAs, y por lo tanto podrían ser factores de confusión en los estudios de circRNA. En los párrafos siguientes explicaremos los datos que respaldan esta propuesta.
Detección de ARNc: Todo Lo Que Brilla No Es Oro
Como se introdujo anteriormente, los circRNAs se forman a través de un evento de empalme no canónico llamado backsplicing. Las transcripciones resultantes de este evento de backsplicing tienen una estructura de bucle cerrado covalentemente sin polaridad de 5 ‘-3′, ni una cola poliadenilada y, lo que es más importante, se caracterizan por la presencia de un orden de exones revuelto en relación con la transcripción lineal (Zhang et al., 2016; Wilusz, 2018). Este orden de exones revuelto se hace evidente en la unión de la pared posterior que conecta una secuencia de 5′ aguas abajo con una secuencia de 3’ aguas arriba. Por lo tanto, todos los algoritmos de detección de ARNc aprovechan la presencia de las uniones de empalme posterior como una característica de diagnóstico para la identificación del ARNc (Figura 1A).
Se han adaptado diferentes métodos para la detección de estas uniones de empalme posterior. Los arrays comerciales que contienen sondas dirigidas a estas regiones con aplicaciones retroactivas se han utilizado ampliamente en estudios de cribado de biomarcadores (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). La validación posterior a menudo también se basa en la amplificación de las uniones backsplicing utilizando cebadores divergentes (Panda y Gorospe, 2018). Muchos otros trabajos han llevado a cabo un análisis de secuenciación de alto rendimiento que supera una de las principales limitaciones de los arrays, lo que permite detectar no solo los ARN anotados, sino también los eventos de circularización de ARN de novo de regiones genómicas donde no se anotaron los ARN en estudios previos. Se han desarrollado varias tuberías bioinformáticas para la detección de circRNAs en conjuntos de datos ARN-Seq, pero todas ellas se basan en la presencia de lecturas que cruzan las uniones de empalme posterior y encontrar la más confiable sigue siendo un desafío para los bioinformáticos (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
Se pueden seguir dos enfoques principales para la detección de ARNs en los datos de ARN-Seq. En primer lugar, muchos estudios ARN-seq de ARN circRNA se basan en muestras tratadas con RNasa R para agotar todos los ARN lineales antes de la secuenciación. Aunque este enfoque está especialmente diseñado para la detección de ARNr, vale la pena señalar que la degradación de la RNasa R es variable, que hay casos raros de ARN lineales resistentes a la RNasa R y ARN sensibles a la RNasa R (Szabo y Salzman, 2016) y que este tratamiento requiere un alto aporte de ARN que podría ser limitante para algunos tejidos. Otros estudios de ARN circular eligen secuenciar ARN total, con agotamiento ribosómico (ribo -) o ARN no poliadenilado (polyA -), donde se pueden encontrar ARN lineales y circulares (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak et al., 2015). Este enfoque evita el uso de RNasa R, que reduce la cantidad de ARN necesaria para la secuenciación y permite estudiar la expresión de otros tipos de ARN a partir del mismo conjunto de datos. Se ha demostrado que con una buena profundidad y calidad de secuenciación y un cuidadoso análisis de datos, se pueden detectar circRNAs verdaderos a partir de la secuenciación total de ARN (Wang et al., 2017), sin embargo, en este segundo enfoque, se necesita una confirmación de circularidad posterior para los circRNAs.
Con el descubrimiento de ARN quiméricos lineales transcritos a partir de ADN circulares, los ARN circulares ya no son las únicas transcripciones con uniones quiméricas. Por lo tanto, es de suma importancia tener en cuenta que mientras que el primer enfoque enriquecerá significativamente la muestra de ARN en circRNAs para que la mayoría de las uniones quiméricas detectadas correspondan a circRNAs verdaderos, el segundo podría sobreestimar el número de transcripciones de circRNAs atribuyendo a los circRNAs la señal proveniente de ambos circRNAs y las transcripciones quiméricas lineales transcritas de DNAs circulares. En consecuencia, teniendo en cuenta la coexistencia de circRNAs y transcripciones quiméricas lineales, la necesidad de pruebas de circularidad y ensayos de funcionalidad gana importancia y se debe tener especial cuidado no solo con los métodos experimentales sino también computacionales para evitar confundir transcripciones quiméricas de ADN circulares con circRNAs formados por backsplicing.
Discusión
El campo de ARNc se encuentra todavía en una etapa temprana, sin embargo, los ARNC ya han demostrado ser moléculas asombrosas, implicadas en muchos procesos, con un gran potencial de biomarcadores y que también pueden cambiar la forma en que entendemos los procesos de transcripción y traducción. Por estas razones, están ganando atención y el campo del circRNA es en este momento uno de los campos de investigación de ARN más activos. Sin embargo, todavía hay muchos conflictos, controversias y preguntas abiertas (Li, 2019) que deben discutirse.
En este informe, y a la luz de los recientes avances en el campo del ADN circular, queremos señalar la transcripción del ADN circular extracromosómico como una de las principales fuentes naturales de transcripciones lineales con señales de empalme posterior que podrían interferir con los datos del circRNA (Møller et al., 2018). A partir de ahora, aparte de los artefactos técnicos, duplicaciones y eventos de transplante que podrían conducir a falsos positivos en la detección del circRNA, también debemos tener en cuenta la existencia de este nuevo tipo de transcripciones quiméricas. Por lo tanto, las pruebas de circularización y los ensayos funcionales son más importantes que nunca.
En cualquier caso, estas transcripciones lineales quiméricas no solo deben considerarse como un mero factor de confusión para los estudios de circRNA. A pesar de las implicaciones técnicas para la caracterización del ARNr, la existencia de estas moléculas de ARN lineal quimérico similares al ARNR que provienen de ECDNAS agrega un nuevo tipo de molécula a la lista cada vez mayor de ARN y expande nuestra visión sobre la complejidad del transcriptoma y su regulación. Por otra parte, estas moléculas de ARN lineal procedentes de ECDNA también podrían presentar funciones similares al circRNA, incluidas las funciones reguladoras o el potencial de ser traducido. Los productos genéticos de las transcripciones de ECDNA podrían contribuir potencialmente al fenotipo de células y tejidos somáticos, según se informa en la levadura (Gresham et al., 2010; Demeke et al., 2015). Sin embargo, en este campo incipiente, se necesitan más datos e investigaciones para comenzar a arañar la superficie del iceberg.
Contribuciones de los autores
LI, IP-L, BR y DO escribieron el artículo.
Financiación
Este estudio ha sido financiado por el Instituto de Salud Carlos III a través del proyecto «PI17 / 00189″(cofinanciado por el Fondo Europeo de Desarrollo Regional / Fondo Social Europeo) «Invertir en tu futuro»). IP-L y BR contaron con el apoyo del Consejo Danés de Investigación Independiente, 6108-00171B y LI contó con el apoyo del Departamento de Educación del Gobierno Vasco .
Conflicto de intereses
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un conflicto de intereses potencial.