Montag, 21. September 2020
Selbst auf dem Gebiet der Genomik, wo alle paar Monate neue Durchbrüche erzielt werden, ist die Fertigstellung des ersten vollständig sequenzierten menschlichen Autosoms eine bedeutende Errungenschaft. Hochpräzise, keine Lücken, keine Fehlverbindungen – nur Chromosom 8 in seiner ganzen Pracht. Es ist eine bemerkenswerte Leistung und wir fühlen uns geehrt, dass PacBio HiFi eine entscheidende Rolle dabei gespielt hat, dies zu erreichen.
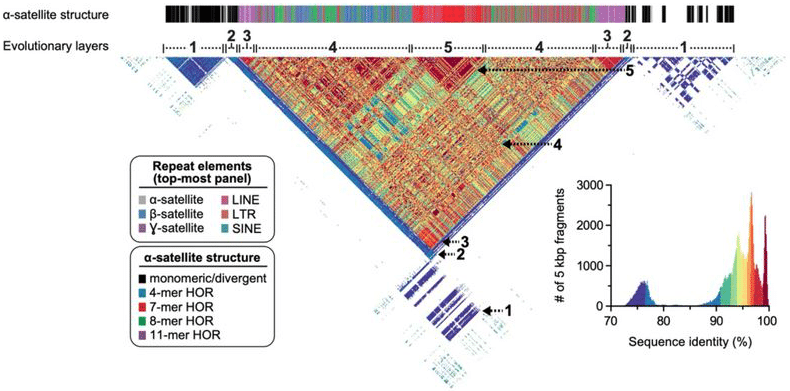
Die vollständige Zentromersequenz von Chromosom 8 zeigt eine Vielfalt von Satellitenwiederholungen und anderen reichlich vorhandenen genomischen Wiederholungen, jetzt mit nahezu perfekter Auflösung auf Basisebene von Ende zu Ende. In: Logsdon, G et al. (2020)
Diese Arbeit wird in einem kürzlich auf bioRxiv veröffentlichten Preprint des Hauptautors Glennis Logsdon (@glennis_logsdon), des leitenden Autors Evan Eichler und ihrer Mitarbeiter im Telomer-zu-Telomer (T2T) -Konsortium beschrieben. Es ist Teil der umfassenderen T2T-Initiative zur Sequenzierung und Assemblierung des ersten wirklich vollständigen menschlichen Genoms und folgt der früheren Veröffentlichung des vollständig sequenzierten X-Chromosoms.
„Seit der Ankündigung der Sequenzierung des menschlichen Genoms vor 20 Jahren sind menschliche Chromosomen aufgrund großer Regionen hochidentischer Wiederholungen innerhalb von Zentromeren, segmentaler Duplikation und der akrozentrischen kurzen Arme von Chromosomen unvollendet geblieben „, stellen die Autoren fest. „Das Aufkommen von Long-Read-Sequenzierungstechnologien und zugehörigen Algorithmen hat es nun erstmals ermöglicht, diese Regionen systematisch aus nativer DNA zusammenzusetzen.“
Chromosom 8 war aufgrund seines überschaubaren Zentromers (zuvor auf 1,5 MB bis 2,2 Mb Länge geschätzt) ein attraktives Ziel für das erste Autosom des T2T. Das Chromosom beherbergt aber auch „eine der strukturell dynamischsten Regionen im menschlichen Genom — den β-Defensin—Gencluster bei 8p23.1 – sowie ein Neozentromer bei 8q21.2, die in den letzten 20 Jahren weitgehend ungelöst waren „, schreiben die Wissenschaftler. Der β-Defensin-Cluster spielt eine Schlüsselrolle bei der angeborenen Immunität, und strukturelle Variationen in dieser Region sind seit langem mit menschlichen Krankheiten verbunden.
Die neue Assembly, die alle fünf zuvor hartnäckigen Lücken im menschlichen Referenzgenom behebt, wurde mit einer cleveren Methode unter Verwendung mehrerer Datensätze erstellt, einschließlich genauer langer Lesevorgänge: „Mehr als die Hälfte der PacBio-Referenzdaten ist in Lesevorgängen von mehr als 17,8 kbp enthalten, mit einer mittleren Genauigkeit von mehr als 99,9%.“ Nach einem Gerüstschritt, der auf Oxford-Nanoporen-Lesevorgängen basiert, wurden aus PacBio-HiFi-Lesevorgängen zusammengesetzte Contigs ausgetauscht, um die Basenpaarauflösung bereitzustellen. „Wir haben die Basenpaargenauigkeit der Sequenzgerüste verbessert, indem wir die rohe ONT-Sequenz durch mehrere konkordante PacBio-HIFI-Contigs ersetzt haben“, berichtet das Team.
Die komplette chr8-Sequenz taktet mit 146 MB und enthält mehr als 3 MB, die in GRCh38 fehlen. Wie Logsdon et al. schreiben Sie: „Das Ergebnis ist eine ganze Chromosomenanordnung mit einer geschätzten Basenpaargenauigkeit von mehr als 99,99%.“
Die Wissenschaftler befassten sich auch mit dem β-Defensin—Gencluster Persnickety, „den wir in einen einzigen 7,06-Mbp-Locus auflösten – wesentlich größer als die 4,56-Mbp-Region im aktuellen menschlichen Referenzgenom“, stellen sie fest. Fast alle diese Sequenzdaten — 99,9934% davon, um genau zu sein – stammten von HiFi-Lesevorgängen. Das komplette Zentromer machte inzwischen 2,08 Mb aus.
Mit dieser schönen Baugruppe in der Hand machte das T2T-Team eine Spritztour. Zunächst validierten sie es mit einer Vielzahl von orthogonalen Werkzeugen wie optischem Mapping. Als nächstes generierten sie Sequenzdaten für die Chromosom-8-Orthologen bei Schimpansen, Makaken und Orang-Utans, um die Sequenzdaten zu vergleichen und die Evolutionsgeschichte des menschlichen Autosoms zu rekonstruieren. „Vergleichende und phylogenetische Analysen zeigen, dass sich die α-Satellitenstruktur höherer Ordnung speziell im Vorfahren des Menschenaffen entwickelt hat und die zentromere Region sich mit einer Schichtsymmetrie entwickelt hat“, schreibt das Team. „Wir schätzen, dass die Mutationsrate der zentromeren Satelliten-DNA um mindestens 2 beschleunigt wird.2-fach, und diese Beschleunigung erstreckt sich über den α-Satelliten höherer Ordnung hinaus in die flankierende Sequenz.“
Schließlich führten die Forscher eine Analyse von Transkripten in voller Länge durch, die mit der Iso-Seq-Methode hergestellt wurden. Dieser Prozess identifizierte „61 proteinkodierende und 33 nichtkodierende Loci, die besser auf diese fertige Chromosom-8-Sequenz als auf GRCh38 abgebildet sind, einschließlich der Entdeckung neuartiger Gene, die auf polymorphe Regionen mit Kopienzahl abgebildet werden“, berichten sie. Zwölf dieser neuen Gene wurden allein in diesem kniffligen β-Defensin-Locus entdeckt.
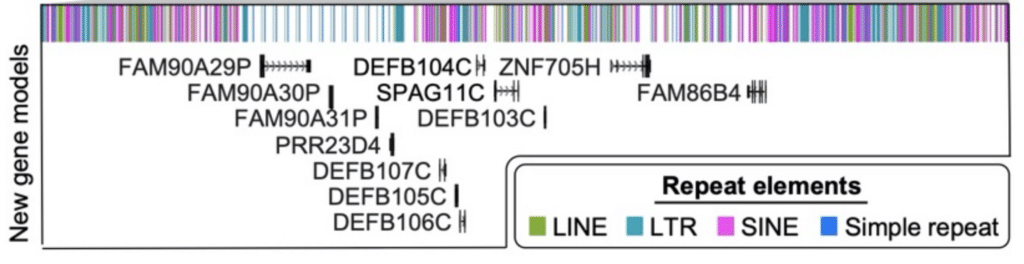
Eine Kombination aus DNA-Genomassemblierung und RNA-Annotation mit Iso-Seq-Daten fügte diese 12 neuen Gene der β-Defensin (DEFB) -Region von Chromosom 8 hinzu. In: Logsdon, G et al. (2020)
Für so viele von uns in der Genomik-Community stellt dieses Papier weit mehr dar als die Sequenz eines einzelnen menschlichen Chromosoms. Es ist eine Aussage darüber, was die Wissenschaft jetzt erreichen kann und wohin uns das in den kommenden Jahren führen könnte. Wie die Autoren zusammenfassten: „Jetzt, da komplexe Regionen wie diese sequenziert und zusammengestellt werden können, wird es wichtig sein, diese Analysen auf andere Zentromere, mehrere Individuen und zusätzliche Arten auszudehnen, um ihre volle Wirkung in Bezug auf genetische Variation und Evolution zu verstehen.“
Sie können weitere Details von Logsdon direkt auf einer kostenlosen Online-Konferenz hören, die vom T2T-Konsortium und dem Human Pangenome Reference Consortium (HPRC) am 22. / 23. Referenten werden neue Einblicke in Chromosom 8 geben und über weitere T2T-Fortschritte auf dem Weg zu einer vollständigen menschlichen Genomassemblierung berichten. Auf der gleichen Veranstaltung wird das HPRC seine komplementären Bemühungen vorstellen, Hunderte von Humangenomen in hoher Qualität zu sequenzieren. Moderatoren sind: Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) und viele mehr. Wir werden dafür an unsere Computer geklebt und hoffen, dass Sie auch die Chance haben, mitzumachen!