Bedeutung
Es gibt ein wachsendes Interesse an circRNAs aufgrund ihrer Implikation in vielen biologischen Prozessen und Krankheiten zusätzlich zu ihrem Biomarkerpotential. Sie werden hauptsächlich durch das Vorhandensein von Reads erkannt, die ihren Backsplicing-Übergang abbilden. Dennoch sind circRNAs nicht mehr die einzigen Transkripte, die einen solchen Übergang enthalten, da neuere Studien gezeigt haben, dass zirkuläre DNAs üblich sind und transkribiert werden können, was zu Transkripten führt, die ein circRNA-Signal nachahmen würden. Daher kann diese neue Art von chimärem Transkript die Art und Weise ändern, in der die circRNA-Analyse durchgeführt wird, und einige der bereits gemeldeten Ergebnisse beeinflussen.
Sind zirkuläre RNAs die einzigen chimären Transkripte?
Zirkuläre RNA (circRNAs) wurde vor einigen Jahren als nicht-kanonisch gespleißte RNA-Formen in verschiedenen Organismen einschließlich des Menschen wiederentdeckt (Salzman et al., 2012; Jeck et al., 2013). Es handelt sich um kovalent geschlossene Transkripte, die durch ein RNA-Rückspleißereignis gebildet werden, bei dem sich ein Spleißdonor eines Downstream-Exons mit einem Upstream-Spleißakzeptor verbindet, was zu kovalent geschlossenen Transkripten führt, die durch das Vorhandensein eines Rückspleißübergangs gekennzeichnet sind, der circRNAs von ihren linearen Gegenstücken unterscheidbar macht (Abbildung 1A) (Zhang et al., 2016; Wilusz, 2018).
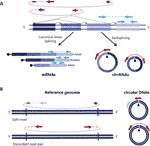
Abbildung 1 Der Nachweis von zirkulärer RNA und DNAs basiert auf Lesevorgängen, die den Übergang überspannen. (A) Backspliced Joining zwischen Exons und canonical Exon Joining werden mit nicht kontinuierlichen bzw. kontinuierlichen Linien dargestellt, die zu circRNAs und mRNAs führen. Lesevorgänge am gepaarten Ende, die die Backspliced-Übergänge überspannen, werden rot angezeigt, und Lesevorgänge am gepaarten Ende, die mit einem linearen Transkript übereinstimmen, werden Blau angezeigt. (B) Ähnlich wie circRNAs werden zirkuläre DNAs (eccDNAs) basierend auf strukturell ausgelesenen Varianten nachgewiesen, die mit einem rot dargestellten Zirkularisierungsereignis übereinstimmen .
Seit ihrer Wiederentdeckung hat die wissenschaftliche Gemeinschaft ihre Aufmerksamkeit auf circRNAs gelenkt und ihre Beteiligung an verschiedenen zellulären Prozessen in Gesundheit und Krankheit (Haque und Harries, 2017), ihre potenzielle Rolle als Biomarker (Abu und Jamal, 2016) und ihre regulatorischen Funktionen (Floris et al., 2016). CircRNAs sind heute im Cytosol und im Zellkern reichlich vorhanden und stabil (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) und wurden auch frei in Biofluiden gefunden (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) und in extrazellulären Vesikeln (Kyoung Mi et al., 2017). Das Biomarkerpotential von circRNAs wurde intensiv untersucht, tatsächlich wurden viele Fall-Kontroll-Studien veröffentlicht, die nach differentiell exprimierten circRNAs suchten, die Biomarker für verschiedene Krankheiten sein könnten. Bisher wurden circRNAs an mehreren Krankheiten einschließlich Krebs beteiligt (Kristensen et al., 2017; Arnaiz et al., 2018), neurologische Störungen (Akhter, 2018), Herz-Kreislauf-Erkrankungen (Aufiero et al., 2019) und immunbedingte Erkrankungen (Iparraguirre et al., 2017; Liu et al., 2019). Gleichzeitig bleiben ihre Biogenese, Eigenschaften, Funktionen und Implikationen für die Humanbiologie als offene Fragen für Forscher auf diesem Gebiet zu verstehen.
Obwohl die Funktion der meisten circRNAs unbekannt bleibt, wurde gezeigt, dass einige circRNAs als microRNA-Schwämme wirken können, die die microRNA-Spiegel und ihre Aktivität regulieren (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Sie sind an der Regulation der Genexpression beteiligt, indem sie die Transkription ihrer Elterngene regulieren und mit den linearen Spleiß- oder Schwammproteinen konkurrieren (Ashwal-Fluss et al., 2014; Li et al., 2018). Interessanterweise haben Ribosomen-Profiling-Studien kürzlich gezeigt, dass circRNAs sowohl in vitro als auch in vivo übersetzt werden können (Legnini et al., 2017; Yang et al., 2017).
Das Hauptmerkmal von circRNAs und verantwortlich für die meisten ihrer besonderen Eigenschaften ist ihre Zirkularität. Daher ist neben dem Nachweis ihrer charakteristischen rückgespleißten Verbindung das Testen der Zirkularität dieser Moleküle einer der Schlüsselpunkte in jeder circRNA-Studie. Dennoch haben viele Studien ihre Entdeckung von circRNAs auf Gesamt-RNA gestützt und könnten dadurch einige lineare chimäre Transkripte als circRNAs interpretiert haben, was zu falsch positiven circRNA-Nachweisen führte. Um dieses Problem zu umgehen, haben die meisten Studien die Zirkularität der Transkripte bestätigt, die durch Gesamt-RNA-seq unter Verwendung von RNase R, Northern Blot oder elektrophoretischen Methoden gefunden wurden (Jeck und Sharpless, 2014). Diese zirkulären Validierungen haben jedoch manchmal auch Transkripte ergeben, die eher linear als zirkulär zu sein scheinen und bestätigen, dass der Nachweis von circRNAs ausgehend von der Gesamt-RNA zu einigen falsch positiven Ergebnissen führen kann. Diese Fehlalarme wurden technischen Artefakten oder Transkripten zugeschrieben, die von ungewöhnlichen Ereignissen wie Exon-Duplikationen oder Transplantationsereignissen abgeleitet wurden (Jeck und Sharpless, 2014; Szabo und Salzman, 2016). Allerdings wurde die Möglichkeit, echte, biologisch aktive und funktionelle lineare Transkripte gefunden zu haben, die eine Sequenz enthalten, die einem Backsplicing-Übergang entspricht (von nun an als chimäre lineare Transkripte bezeichnet), etwas übersehen, da eine Quelle solcher linearer RNA für gesunde Zellen nicht bekannt ist.
Zirkuläre DNAs als Quelle chimärer linearer Transkripte
Der größte Teil des menschlichen Genoms ist in linearen Chromosomen organisiert, einige Ausnahmen wurden jedoch lange akzeptiert, wie mitochondriale DNA und Chromosomenaberrationen wie DNA-Kreise, die Onkogene tragen (z. B. Doppelbindungen) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) und Ringchromosomen (Tümer et al., 2004). Erst vor kurzem konnten verschiedene zirkuläre DNAs wie microDNAs (Shibata et al., 2012) oder extrachromosomale zirkuläre DNAs (eccDNAs) wurden auch aus großen Teilen verschiedener eukaryotischer Genome einschließlich Mensch und Hefe gefunden (Møller et al., 2015; Köhler et al., 2017; Møller et al., 2018).
Zirkuläre DNAs werden gebildet, wenn zwei Enden einer linearen DNA miteinander verbunden sind, was zu einem Übergang führt, der dem rückgespleißten Übergang auf circRNAs ähnelt, der allgemein als Breakpoint Junction bezeichnet wird und basierend auf strukturell gelesenen Varianten im Einklang mit einem Zirkularisierungsereignis nachgewiesen wird (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (Abbildung 1B). Sie reichen normalerweise von hundert Basen bis zu Megabase-Kreisen und können vollständige Exons und Gene enthalten (Shibata et al ., 2012; Møller et al., 2015; Köhler et al., 2017; Turner et al., 2017; Møller et al., 2018) und während einige Regionen des Genoms häufiger auf zirkulärer DNA zu finden sind (Sinclair und Guarente, 1997;; Møller et al., 2016; Turner et al., 2017; Møller et al., 2018) scheinen die meisten zirkulären DNA zufällig aufzutreten (Shibata et al., 2012; Møller et al., 2015; Köhler et al., 2017; Møller et al., 2018).
Interessanterweise haben Møller et al. identifizierte Tausende von eccDNAs in Leukozyten und Muskelzellen in gesunden Kontrollen. Mit der Idee zu untersuchen, ob eccDNAs transkribiert werden können, wurde auch eine mRNA-Bibliothek aus Muskelgewebe sequenziert und auf Transkriptionsereignisse über den Breakpoint-Übergang der detektierten eccDNA analysiert, wobei mehrere Übereinstimmungen gefunden wurden (Møller et al., 2018). Dieser Befund legt nahe, dass zirkuläre DNA in gesundem Gewebe transkribiert wird, was zu linearen und polyadenylierten Transkripten führt, die eine Sequenz tragen, die der Backsplicing-Sequenz von circRNAs entspricht (Møller et al., 2018) (Abbildung 2).
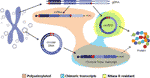
Abbildung 2 Grafische Darstellung verschiedener Transkripte, die entweder aus genomischer DNA oder zirkulärer DNA entstanden sind. Exons sind lila gefärbt und die rückgespleißten Übergänge oder chimären Übergänge sind rot dargestellt. Polyadenylierte, chimäre Verbindungen enthaltende und RNase R-resistente Transkripte sind orange, blau bzw. gelb hervorgehoben.
Die Transkriptionsnachweise für zirkuläre DNAs zusammen mit ihrer Häufigkeit lassen vermuten, dass zirkuläre DNAs eine natürliche Quelle für eine beträchtliche Menge linearer RNAs sein könnten, die chimäre Übergänge tragen. In vielen Fällen sind diese chimären Übergänge möglicherweise nicht von den Backsplicing-Übergängen von circRNAs zu unterscheiden, und daher können sie Störfaktoren in circRNA-Studien sein. In den folgenden Abschnitten werden wir die Daten erläutern, die diesen Vorschlag unterstützen.
circRNA-Nachweis: Alles, was glänzt, ist nicht Gold
Wie bereits erwähnt, werden circRNAs durch ein nicht-kanonisches Spleißereignis gebildet, das als Backsplicing bezeichnet wird. Transkripte, die aus diesem Backsplicing-Ereignis resultieren, haben eine kovalent geschlossene Schleifenstruktur mit weder 5′-3′ Polarität noch einem polyadenylierten Schwanz und was noch wichtiger ist, sie sind durch das Vorhandensein einer verschlüsselten Exonordnung relativ zum linearen Transkript gekennzeichnet (Zhang et al., 2016; Wilusz, 2018). Diese verschlüsselte Exonreihenfolge wird in der rückgespleißten Verbindung deutlich, die eine 5′-Downstream-Sequenz mit einer Upstream-3′-Sequenz verbindet. Somit nutzen alle circRNA-Detektionsalgorithmen das Vorhandensein der rückgespleißten Übergänge als diagnostisches Merkmal für die Identifizierung von circRNA (Abbildung 1A).
Für die Detektion dieser Back-spliced Junctions wurden verschiedene Methoden adaptiert. Kommerzielle Arrays, die Sonden enthalten, die auf diese rückgespleißten Regionen abzielen, wurden in Biomarker-Screening-Studien häufig verwendet (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). Die anschließende Validierung basiert häufig auch auf der Amplifikation der Backsplicing-Übergänge unter Verwendung divergenter Primer (Panda und Gorospe, 2018). Viele andere Arbeiten haben eine Hochdurchsatz-Sequenzierungsanalyse durchgeführt, die eine der Hauptbeschränkungen der Arrays überwindet, die es ermöglicht, nicht nur die annotierten circRNAs, sondern auch De-novo-RNA-Zirkularisierungsereignisse aus genomischen Regionen zu erkennen, in denen in früheren Studien keine circRNA annotiert wurde. Für den Nachweis von circRNAs in RNA-Seq-Datensätzen wurden mehrere bioinformatische Pipelines entwickelt, aber alle basieren auf dem Vorhandensein von Reads, die die Back-Splicing-Übergänge kreuzen, und das Finden der zuverlässigsten ist für Bioinformatiker immer noch eine Herausforderung (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
Für den Nachweis von circRNAs in RNA-Seq-Daten können zwei Hauptansätze verfolgt werden. Erstens basieren viele circRNA-RNA-seq-Studien auf mit RNase R behandelten Proben, um alle linearen RNAs vor der Sequenzierung abzubauen. Obwohl dieser Ansatz speziell für den circRNA-Nachweis entwickelt wurde, ist anzumerken, dass der RNase-R-Abbau variabel ist, dass es seltene Fälle von RNase-R-resistenten linearen RNAs und RNase-R-sensitiven circRNAs gibt (Szabo und Salzman, 2016) und dass diese Behandlung einen hohen RNA-Input erfordert, der für einige Gewebe limitierend sein könnte. Andere circRNA-Studien sequenzieren entweder totale, ribosomal abgereicherte (Ribo-) oder nicht polyadenylierte (polyA-) RNA, wobei sowohl lineare als auch zirkuläre RNAs gefunden werden können (Salzman et al., 2012; Memczak et al., 2013; Broad et al., 2015; Lu et al., 2015; In: Memczak et al., 2015). Dieser Ansatz vermeidet die Verwendung von RNase R, wodurch die für die Sequenzierung erforderliche RNA-Menge reduziert wird und die Expression anderer Arten von RNAs aus demselben Datensatz untersucht werden kann. Es wurde gezeigt, dass mit einer guten Sequenzierungstiefe und -qualität und einer sorgfältigen Datenanalyse echte circRNAs aus der Gesamt-RNA-Sequenzierung nachgewiesen werden können (Wang et al., 2017), in diesem zweiten Ansatz ist jedoch eine spätere Bestätigung der Zirkularität für circRNAs erforderlich.
Mit der Entdeckung linearer chimärer RNAs, die aus zirkulären DNAs transkribiert wurden, sind circRNAs nicht mehr die einzigen Transkripte mit chimären Übergängen. Daher ist es von größter Bedeutung zu beachten, dass der erste Ansatz die RNA-Probe in circRNAs signifikant anreichert, so dass die meisten der nachgewiesenen chimären Übergänge echten circRNAs entsprechen, der zweite Ansatz könnte die Anzahl der circRNA-Transkripte überschätzen, indem er circRNAs das Signal zuschreibt, das sowohl von circRNAs als auch von den linearen chimären Transkripten kommt, die von zirkulären DNAs transkribiert werden. Folglich, unter Berücksichtigung der Koexistenz von circRNAs und linearen chimären Transkripten, Die Notwendigkeit von Zirkularitätstests und Funktionalitätstests gewinnt an Bedeutung und besondere Sorgfalt sollte nicht nur in Bezug auf experimentelle, sondern auch auf rechnerische Methoden angewendet werden, um zu vermeiden, dass chimäre Transkripte aus zirkulären DNAs mit circRNAs verwechselt werden, die durch Backsplicing gebildet werden.
Diskussion
Das circRNA-Feld befindet sich noch in einem frühen Stadium, aber circRNAs haben sich bereits als erstaunliche Moleküle erwiesen, die an vielen Prozessen beteiligt sind und ein großes Biomarkerpotenzial haben und das kann auch das Verständnis der Transkriptions- und Translationsprozesse verändern. Aus diesen Gründen gewinnen sie an Aufmerksamkeit und das circRNA-Feld ist derzeit eines der aktivsten RNA-Forschungsfelder. Es gibt jedoch noch viele Konflikte, Kontroversen und offene Fragen (Li, 2019), die diskutiert werden müssen.
In diesem Bericht und angesichts der jüngsten Fortschritte auf dem Gebiet der zirkulären DNA möchten wir auf die Transkription aus extrachromosomaler zirkulärer DNA als eine der wichtigsten natürlichen Quellen für lineare Transkripte mit rückgespleißten Signalen hinweisen, die circRNA-Daten stören könnten (Møller et al., 2018). Von nun an sollten wir neben den technischen Artefakten, Duplikationen und Transplantationsereignissen, die zu falsch positiven Ergebnissen beim circRNA-Nachweis führen könnten, auch die Existenz dieser neuen Art von chimären Transkripten berücksichtigen. Zirkularisierungstests und funktionelle Assays sind daher wichtiger denn je.
In jedem Fall sollten diese chimären linearen Transkripte nicht nur als bloßer Störfaktor für circRNA-Studien betrachtet werden. Trotz der technischen Implikationen für die circRNA-Charakterisierung fügt die Existenz dieser circRNA-ähnlichen chimären linearen RNA-Moleküle, die aus eccDNAs stammen, der ständig wachsenden Liste von RNAs einen neuen Molekültyp hinzu und erweitert unsere Vision über die Komplexität des Transkriptoms und seiner Regulation. Darüber hinaus könnten diese linearen RNA-Moleküle, die aus der eccDNA stammen, auch ähnliche Funktionen wie die circRNA aufweisen, einschließlich der regulatorischen Funktionen oder des Translationspotenzials. Genprodukte aus eccDNA-Transkripten könnten potenziell zum Phänotyp somatischer Zellen und Gewebe beitragen, wie in Hefe berichtet (Gresham et al., 2010; Demeke et al., 2015). In diesem aufstrebenden Bereich sind jedoch mehr Daten und Forschungen erforderlich, um an der Oberfläche des Eisbergs zu kratzen.
Autorenbeiträge
LI, IP-L, BR und DO schrieben das Papier.
Finanzierung
Diese Studie wurde vom Instituto de Salud Carlos III im Rahmen des Projekts „PI17 /00189“ (kofinanziert durch den Europäischen Fonds für regionale Entwicklung / Europäischen Sozialfonds) „Investing in your future“) finanziert. IP-L und BR wurden vom dänischen Rat für unabhängige Forschung, 6108-00171B und LI vom Bildungsministerium der baskischen Regierung unterstützt .
Interessenkonflikt
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.