maandag 21 September 2020
zelfs op het gebied van de Genomica, waar om de paar maanden nieuwe doorbraken plaatsvinden, is de voltooiing van het allereerste volledig gesequenced menselijk autosoom een gedenkwaardige prestatie. Zeer nauwkeurig, geen gaten, geen mis-joins – gewoon chromosoom 8 in al zijn glorie. Het is een opmerkelijke prestatie en we zijn vereerd dat PacBio HiFi reads speelde een centrale rol in het helpen om het te bereiken.
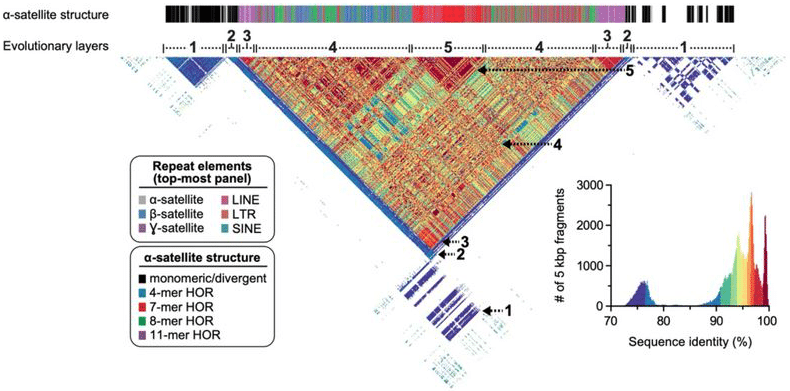
de volledige centromeeropeenvolging van chromosoom 8 toont een diversiteit van satelliet herhaalt en andere overvloedige genomic herhaalt, nu met dichtbij perfecte basis-niveau resolutie van eind aan eind. Logsdon, G et al. (2020)
dit werk wordt beschreven in een preprint die onlangs in bioRxiv is geplaatst door hoofdauteur Glennis Logsdon (@glennis_logsdon), senior auteur Evan Eichler, en hun medewerkers in het Consortium Telomere-to-Telomere (T2T). Het maakt deel uit van het bredere initiatief T2T om het eerste Echt volledige menselijke genoom te rangschikken en te assembleren en volgt de eerdere versie van het volledig gesequenced chromosoom van X.
” sinds de aankondiging van de sequencing van het menselijk genoom 20 jaar geleden, zijn menselijke chromosomen onafgewerkt gebleven vanwege grote gebieden van sterk identieke herhalingen binnen centromeren, segmentale duplicatie en de acrocentrische korte armen van chromosomen,” merken de auteurs op. “De komst van long-read sequencing technologieën en bijbehorende algoritmen hebben het nu mogelijk gemaakt om deze regio’ s systematisch te assembleren uit native DNA voor de eerste keer.”
chromosoom 8 maakte een aantrekkelijk doelwit voor het eerste autosoom van de T2T vanwege zijn beheersbare centromeer (eerder geschat op 1,5 MB tot 2,2 MB lang). Maar het chromosoom is ook de thuisbasis van ” een van de meest structureel dynamische gebieden in het menselijk genoom—De β-defensin gencluster gelegen op 8p23.1—evenals een neocentromere gelegen op 8q21.2, die grotendeels onopgelost voor de laatste 20 jaar, ” de wetenschappers schrijven. De β-defensincluster speelt een sleutelrol in aangeboren immuniteit en structurele variatie in dit gebied is al lang betrokken bij menselijke ziekte.
de nieuwe assemblage, die alle vijf de voorheen hardnekkige hiaten in het menselijk referentiegenoom aanpakt, werd gebouwd met een slimme methode met behulp van verschillende datasets, waaronder nauwkeurige lange reads: “meer dan de helft van de PacBio HiFi-gegevens is opgenomen in reads groter dan 17,8 kbp, met een mediane nauwkeurigheid van meer dan 99,9%.”Na een steigerstap op basis van Oxford Nanopore reads, werden contigs samengesteld uit Pacb HiFi reads geruild om de basispaar resolutie te bieden. “We hebben de nauwkeurigheid van het basispaar van de sequence scaffolds verbeterd door de raw ONT sequence te vervangen door verschillende Concordante PacBio HiFi contigs,” meldt het team.
de volledige chr8-reeks klokt 146 Mb en bevat meer dan 3 MB die ontbreekt in GRCh38. As Logsdon et al. schrijf: “het resultaat is een geheel-chromosoom assemblage met een geschatte base-pair nauwkeurigheid van meer dan 99,99%.”
de wetenschappers ook aangepakt dat persnickety β-defensin gen cluster, ” die we opgelost in een enkele 7.06 Mbp locus-aanzienlijk groter dan de 4,56 MBP regio in het huidige menselijke referentie genoom,” merken ze op. Bijna al die sequentiegegevens — 99,9934% ervan, om precies te zijn — kwamen van HiFi reads. De volledige centromeer, ondertussen, goed voor 2,08 Mb.
met deze prachtige assemblage in de hand, ging het T2T-team er een rondje mee rijden. Eerst valideerden ze het met een groot aantal orthogonale hulpmiddelen, zoals optische mapping. Vervolgens produceerden ze HiFi-gegevens voor de chromosoom 8-orthologen in chimpansee, makaak en orang-oetan om de sequentiegegevens te vergelijken en de evolutionaire geschiedenis van het menselijk autosoom te reconstrueren. “Vergelijkende en fylogenetische analyses tonen aan dat de hogere orde α-satelliet structuur specifiek geëvolueerd in de grote aap voorouder, en de centromere regio geëvolueerd met een gelaagde symmetrie,” het team schrijft. “We schatten dat de mutatiesnelheid van centromerische satelliet DNA is versneld ten minste 2.2-voudig, en deze versnelling strekt zich uit voorbij de hogere orde α-satelliet in de flankerende sequentie.”
ten slotte voerden de onderzoekers een analyse uit van volledige transcripten geproduceerd met de Iso-Seq methode. Dat proces identificeerde ” 61 eiwit-codeert en 33 noncoding loci die beter aan deze gebeëindigde chromosoom 8 opeenvolging dan aan GRCh38 in kaart brengen, met inbegrip van de ontdekking van nieuwe genen die aan de polymorfe gebieden van het exemplaaraantal in kaart brengen,” melden zij. Twaalf van deze nieuwe genen werden ontdekt in die lastige β-defensin locus alleen.
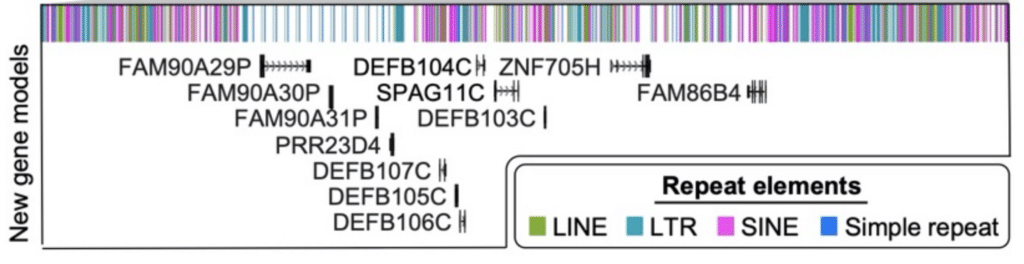
een combinatie van de assemblage van het hifi-genoom en de annotatie van RNA met Iso-Seq gegevens voegde deze 12 nieuwe genen aan het gebied van β-defensin (DEFB) van chromosoom 8 toe. Logsdon, G et al. (2020)
voor zo velen van ons in de Genomica-Gemeenschap vertegenwoordigt dit artikel veel meer dan de sequentie van een enkel menselijk chromosoom. Het is een statement over wat de wetenschap nu kan bereiken, en waar dat ons in de komende jaren kan leiden. Zoals de auteurs samengevat: “Nu complexe gebieden zoals deze kunnen worden gesequenced en geassembleerd, zal het belangrijk zijn om deze analyses uit te breiden naar andere centromeren, meerdere individuen en extra soorten om hun volledige impact met betrekking tot genetische variatie en evolutie te begrijpen.”
u kunt meer details van Logsdon rechtstreeks horen op een gratis online conferentie die wordt georganiseerd door het T2T Consortium en het Human Pangenome Reference Consortium (HPRC) op 22/23 September. Sprekers zullen nieuwe inzichten bieden over chromosoom 8 en verslag uitbrengen over verdere T2T vooruitgang naar een complete menselijke genoom assemblage. Op hetzelfde evenement presenteert het HPRC zijn complementaire inspanning om honderden menselijke genomen te sequencen tot hoge kwaliteit. Presentatoren zijn: Karen Miga (@khmiga), Eric Groen (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graven-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) en nog veel meer. We zullen vastgelijmd aan onze computers voor het en we hopen dat u een kans om ook mee te doen!