segunda-feira, 21 de setembro de 2020
mesmo no campo da genómica, onde novos avanços ocorrem a cada poucos meses, a conclusão do primeiro autossoma humano totalmente sequenciado é uma conquista momentosa. Altamente preciso, sem falhas, sem falhas-apenas cromossomas 8 em toda a sua glória. É um feito notável e estamos honrados por PacBio HiFi reads ter desempenhado um papel fundamental em ajudar a alcançá-lo.
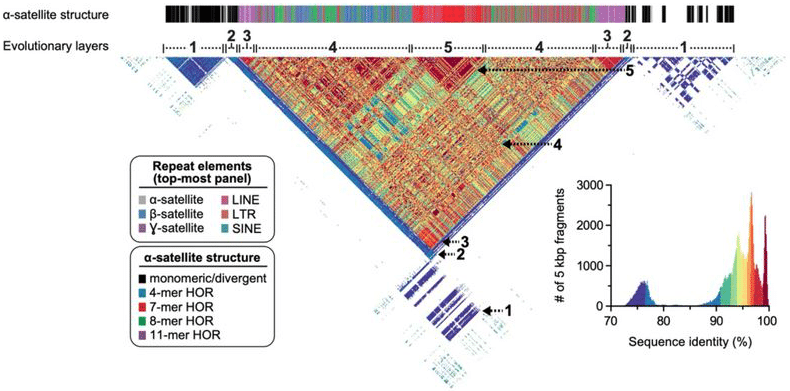
a sequência completa de centrômero do cromossomo 8 mostra uma diversidade de repetições de satélite e outras repetições genômicas abundantes, agora com uma resolução quase perfeita de nível de base de ponta a ponta. Logsdon, G et al. (2020)
Este trabalho é descrito em um preprint postou recentemente para bioRxiv do autor principal Glennis Logsdon (@glennis_logsdon), autor sênior do Evan Eichler, e seus colaboradores no Telômero-para-Telômero (T2T) Consórcio. Faz parte da iniciativa mais ampla T2T para sequenciar e montar o primeiro genoma humano verdadeiramente completo e segue a liberação anterior do cromossomo X totalmente sequenciado.
“desde o anúncio da sequenciação do genoma humano há 20 anos, os cromossomas humanos têm permanecido inacabados devido a grandes regiões de repetições altamente idênticas localizadas dentro de centrómeros, duplicação segmental e os braços curtos acrocêntricos dos cromossomas”, observam os autores. “O advento de tecnologias de sequenciamento de longa leitura e algoritmos associados tornaram agora possível a montagem sistemática dessas regiões a partir do DNA nativo pela primeira vez.”
o Cromossoma 8 fez um alvo atraente para o primeiro autossomo do T2T devido ao seu centrômero gerenciável (anteriormente estimado em 1,5 Mb a 2,2 Mb de comprimento). Mas o cromossomo também é o lar de ” uma das regiões estruturalmente mais dinâmicas do genoma humano—o aglomerado genético de β-defensina localizado em 8p23.1—, bem como um neocentromere localizado em 8q21.2, que têm estado em grande parte por resolver nos últimos 20 anos”, escrevem os cientistas. O cluster de β-defensina desempenha um papel fundamental na imunidade inata e a variação estrutural nesta região tem sido há muito implicada na doença humana.
a nova montagem, que aborda todas as cinco lacunas anteriormente intratáveis no genoma de referência humana, foi construída com um método inteligente usando vários conjuntos de dados, incluindo leituras longas e precisas: “mais da metade dos dados do PacBio HiFi está contida em leituras superiores a 17,8 kbp, com uma precisão mediana superior a 99,9%.”Depois de um passo de andaime baseado em Oxford Nanopore reads, contigs montados a partir de PacBio HiFi reads foram trocados para fornecer a resolução do par base. “Nós melhoramos a precisão do par de base dos andaimes de seqüência, substituindo a sequência ONT raw por vários concordantes PacBio HiFi contigs”, relata a equipe.
The complete chr8 sequence clocks in at 146 Mb and includes more than 3 Mb missing from GRCh38. As Logsdon et al. write, ” The result is a whole-chromosome assembly with an estimated base-pair accuracy exceeding 99.99%.”
Os cientistas também abordou que persnickety β-defensin gene cluster”, que nós resolvidas em um único 7.06 Mbp locus—substancialmente maior do que o 4.56 Mbp região da corrente humana de referência do genoma,” eles observação. Quase todos os dados da sequência-99.9934%, para ser preciso — vieram de leituras HiFi. O centrômero completo, entretanto, representava 2,08 Mb.Com esta bela montagem na mão, a equipa T2T levou-a para dar uma volta. Primeiro, validaram-no com uma série de ferramentas ortogonais, como o mapeamento óptico. Em seguida, eles geraram dados HiFi para o cromossomo 8 orthologs em chimpanzé, macaque e orangotango para comparar os dados de sequência e reconstruir a história evolutiva do autossomo humano. “Análises comparativas e filogenéticas mostram que a estrutura α-satélite de ordem superior evoluiu especificamente no ancestral do Grande macaco, e a região centromérica evoluiu com uma simetria em camadas”, Escreve a equipe. “Estimamos que a taxa de mutação do DNA de satélite centromérico é acelerada pelo menos 2.2 vezes, e esta aceleração estende-se para além do satélite α de ordem superior para a sequência de flanco.”
Finally, the researchers performed an analysis of full-length transcripts produced with the Iso-Seq method. Esse processo identificou “61 códigos de proteínas e 33 loci não codificantes que mapeiam melhor a esta sequência final do cromossomo 8 do que a GRCh38, incluindo a descoberta de novos genes mapeando para copiar as regiões polimórficas de número”, relatam. Doze destes novos genes foram descobertos apenas nesse complexo locus de β-defensina.
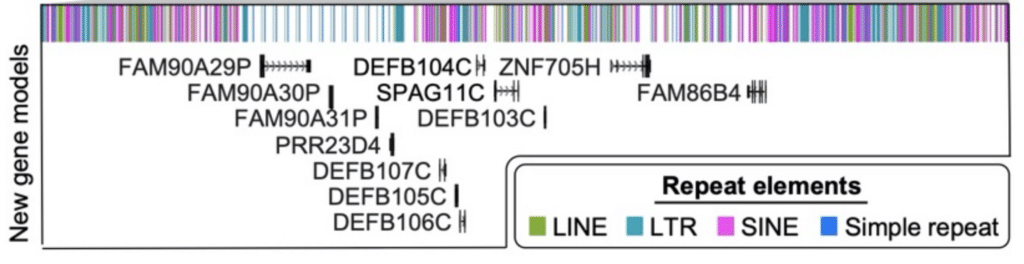
uma combinação do conjunto do genoma HiFi e da anotação do ARN com os dados Iso-Seq adicionou estes 12 novos genes à região de β-defensina (DEFB) do cromossoma 8. Logsdon, G et al. (2020)
para muitos de nós na comunidade genómica, este artigo representa muito mais do que a sequência de um único cromossoma humano. É uma declaração sobre o que a ciência pode realizar agora, e onde isso pode nos levar nos próximos anos. Como os autores resumiram: “Agora que regiões complexas como estas podem ser sequenciadas e montadas, será importante estender essas análises a outros centrômeros, múltiplos indivíduos e espécies adicionais para entender seu pleno impacto com relação à variação genética e evolução.”
você pode ouvir mais detalhes da Logsdon diretamente em uma conferência online gratuita co-organizada pelo Consórcio T2T e Human Pangenome Reference Consortium (HPRC) em 22 de setembro. Os oradores apresentarão novas ideias sobre o cromossoma 8 e apresentarão um relatório sobre os novos progressos do T2T no sentido de uma montagem completa do genoma humano. No mesmo evento, o HPRC apresentará o seu esforço complementar para sequenciar centenas de genomas humanos a alta qualidade. Apresentadores incluem: Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Sepulturas-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Marcos Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) e muitos mais. Estaremos colados aos nossos computadores para isso e esperamos que você tenha uma chance de se juntar também!