Lunedì 21 settembre 2020
Anche nel campo della genomica, dove nuove scoperte si verificano ogni pochi mesi, il completamento del primo autosoma umano completamente sequenziato è un risultato epocale. Altamente preciso, senza lacune, senza mis-join – solo cromosoma 8 in tutta la sua gloria. È un’impresa notevole e siamo onorati che le letture di PacBio HiFi abbiano avuto un ruolo fondamentale nell’aiutare a raggiungerlo.
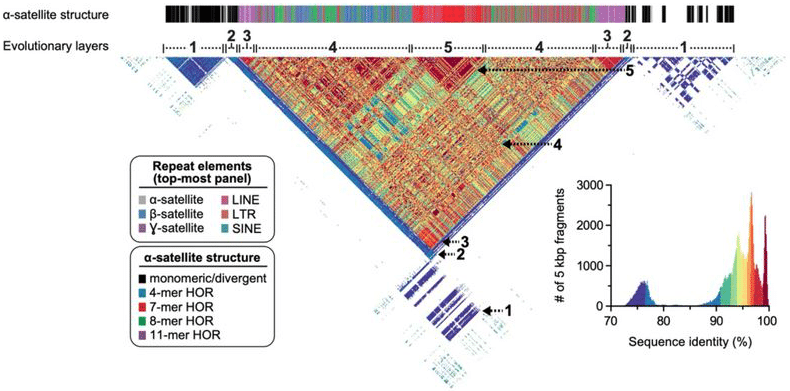
La sequenza centromerica completa del cromosoma 8 mostra una diversità di ripetizioni satellitari e altre abbondanti ripetizioni genomiche, ora con una risoluzione quasi perfetta a livello di base da un capo all’altro. Logsdon, G et al. (2020)
Questo lavoro è descritto in un preprint recentemente pubblicato su bioRxiv dall’autore principale Glennis Logsdon (@glennis_logsdon), dall’autore senior Evan Eichler e dai loro collaboratori nel Consorzio Telomere-to-Telomere (T2T). Fa parte della più ampia iniziativa T2T per sequenziare e assemblare il primo genoma umano veramente completo e segue il rilascio precedente del cromosoma X completamente sequenziato.
“Dall’annuncio del sequenziamento del genoma umano 20 anni fa, i cromosomi umani sono rimasti incompiuti a causa di ampie regioni di ripetizioni altamente identiche situate all’interno dei centromeri, della duplicazione segmentale e dei bracci corti acrocentrici dei cromosomi”, notano gli autori. “L’avvento di tecnologie di sequenziamento a lunga lettura e algoritmi associati hanno ora reso possibile assemblare sistematicamente queste regioni dal DNA nativo per la prima volta.”
Il cromosoma 8 ha reso un bersaglio attraente per il primo autosoma del T2T grazie al suo centromero gestibile (precedentemente stimato da 1,5 Mb a 2,2 Mb di lunghezza). Ma il cromosoma ospita anche “una delle regioni strutturalmente più dinamiche del genoma umano—il cluster del gene β-defensin situato a 8p23.1—così come un neocentromero situato a 8q21.2, che sono stati in gran parte irrisolti negli ultimi 20 anni”, scrivono gli scienziati. Il cluster β-defensin svolge un ruolo chiave nell’immunità innata e la variazione strutturale in questa regione è stata a lungo implicata nella malattia umana.
Il nuovo assembly, che risolve tutte e cinque le lacune precedentemente intrattabili nel genoma umano di riferimento, è stato costruito con un metodo intelligente utilizzando diversi set di dati, tra cui accurate letture lunghe: “Più della metà dei dati PacBio HiFi è contenuta in letture superiori a 17,8 kbp, con una precisione mediana superiore al 99,9%.”Dopo una fase di impalcatura basata su Oxford Nanopore reads, i contig assemblati da PacBio HiFi reads sono stati scambiati per fornire la risoluzione della coppia di basi. “Abbiamo migliorato la precisione della coppia base degli scaffold di sequenza sostituendo la sequenza ONT grezza con diversi contig PACBIO HiFi concordanti”, riferisce il team.
La sequenza chr8 completa si blocca a 146 Mb e include più di 3 Mb mancanti da GRCh38. Come Logsdon et al. scrivi: “Il risultato è un assemblaggio cromosomico intero con una precisione stimata della coppia di basi superiore al 99,99%.”
Gli scienziati hanno anche affrontato quel cluster di geni β-defensin persnickety, “che abbiamo risolto in un singolo locus 7.06 Mbp—sostanzialmente più grande della regione 4.56 Mbp nell’attuale genoma umano di riferimento”, notano. Quasi tutti i dati di quella sequenza — il 99,9934%, per essere precisi-provenivano da letture HiFi. Il centromero completo, nel frattempo, rappresentava 2,08 Mb.
Con questo bellissimo assemblaggio in mano, il team T2T lo ha tirato fuori per un giro. In primo luogo, lo hanno convalidato con una serie di strumenti ortogonali, come la mappatura ottica. Successivamente, hanno generato dati HiFi per gli ortologi del cromosoma 8 in scimpanzé, macaco e orangutan per confrontare i dati di sequenza e ricostruire la storia evolutiva dell’autosoma umano. “Analisi comparative e filogenetiche mostrano che la struttura α-satellite di ordine superiore si è evoluta specificamente nell’antenato della grande scimmia e la regione centromerica si è evoluta con una simmetria stratificata”, scrive il team. “Stimiamo che il tasso di mutazione del DNA satellite centromerico sia accelerato almeno 2.2 volte, e questa accelerazione si estende oltre il satellite α di ordine superiore nella sequenza di fiancheggiamento.”
Infine, i ricercatori hanno eseguito un’analisi delle trascrizioni a lunghezza intera prodotte con il metodo Iso-Seq. Quel processo ha identificato “61 loci codificanti proteine e 33 non codificanti che mappano meglio questa sequenza cromosomica 8 finita rispetto a GRCh38, inclusa la scoperta di nuovi geni che mappano per copiare il numero di regioni polimorfiche”, riferiscono. Dodici di questi nuovi geni sono stati scoperti solo in quel complicato locus β-defensin.
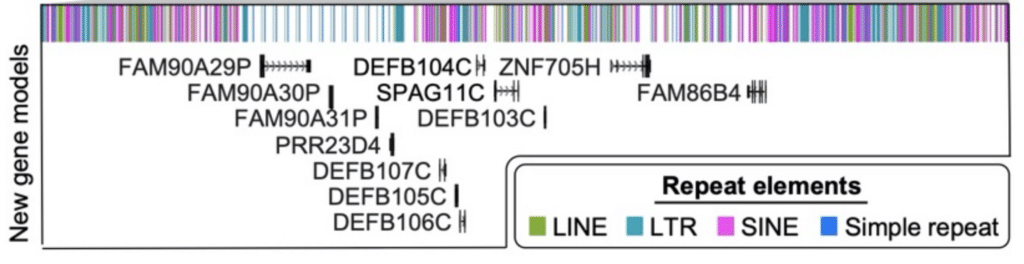
Una combinazione di assemblaggio del genoma HiFi e annotazione dell’RNA con dati Iso-Seq ha aggiunto questi 12 nuovi geni alla regione β-defensina (DEFB) del cromosoma 8. Logsdon, G et al. (2020)
Per molti di noi nella comunità genomica, questo documento rappresenta molto di più della sequenza di un singolo cromosoma umano. È una dichiarazione su ciò che la scienza può realizzare ora e dove ciò potrebbe condurci negli anni a venire. Come gli autori hanno riassunto: “Ora che regioni complesse come queste possono essere sequenziate e assemblate, sarà importante estendere queste analisi ad altri centromeri, più individui e altre specie per comprendere il loro pieno impatto rispetto alla variazione e all’evoluzione genetica.”
Puoi sentire maggiori dettagli da Logsdon direttamente in una conferenza online gratuita co-ospitata dal Consorzio T2T e Human Pangenome Reference Consortium (HPRC) il 22/23 settembre. I relatori offriranno nuove informazioni sul cromosoma 8 e riferiranno su ulteriori progressi T2T verso un assemblaggio completo del genoma umano. Allo stesso evento, l’HPRC presenterà il suo sforzo complementare per sequenziare centinaia di genomi umani ad alta qualità. I relatori sono: Karen Miga (@khmiga), Eric Verde (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Tombe-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Marco Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) e molti altri. Saremo incollati ai nostri computer per questo e speriamo che avrete la possibilità di aderire pure!