måndag 21 September 2020
även inom genomik där nya genombrott inträffar med några månaders mellanrum är slutförandet av den allra första fullständigt sekvenserade mänskliga autosomen en betydelsefull prestation. Mycket exakt, inga luckor, inga felkopplingar – bara kromosom 8 i all sin härlighet. Det är en anmärkningsvärd prestation och vi är hedrade att PacBio HiFi reads spelade en central roll för att hjälpa till att uppnå det.
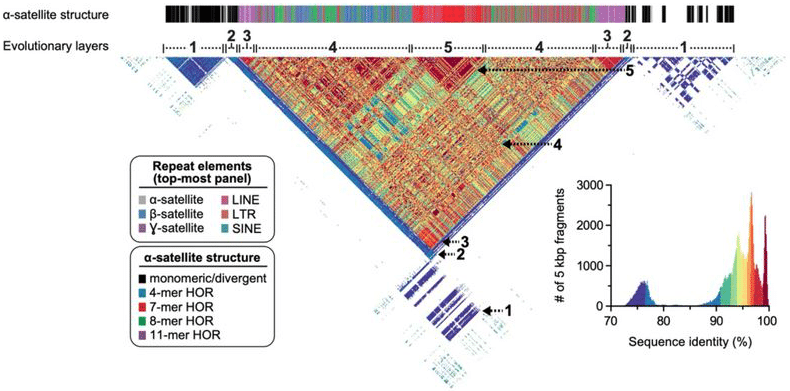
den kompletta centromersekvensen av kromosom 8 visar en mångfald av satellitupprepningar och andra rikliga genomiska upprepningar, nu med nästan perfekt basnivåupplösning från slut till slut. Logsdon, G et al. (2020)
detta arbete beskrivs i ett förtryck som nyligen publicerats till bioRxiv från huvudförfattaren Glennis Logsdon (@glennis_logsdon), seniorförfattaren Evan Eichler och deras medarbetare i konsortiet Telomere-to-Telomere (T2T). Det är en del av det bredare T2T-initiativet att sekvensera och montera det första verkligt kompletta mänskliga genomet och följer den tidigare frisättningen av den fullständigt sekvenserade X-kromosomen.
”sedan tillkännagivandet av sekvenseringen av det mänskliga genomet för 20 år sedan har mänskliga kromosomer förblivit oavslutade på grund av stora regioner med mycket identiska upprepningar belägna inom centromerer, segmentell dubbelarbete och de akrocentriska korta armarna av kromosomer”, noterar författarna. ”Tillkomsten av långläst sekvenseringsteknik och tillhörande algoritmer har nu gjort det möjligt att systematiskt montera dessa regioner från nativt DNA för första gången.”
kromosom 8 gjorde ett attraktivt mål för T2T: s första autosom på grund av dess hanterbara centromer (tidigare uppskattad till 1, 5 Mb till 2, 2 Mb lång). Men kromosomen är också hem för ” en av de mest strukturellt dynamiska regionerna i det mänskliga genomet—genklyftan för hCG-defensin som ligger vid 8p23.1—liksom en neocentromer som ligger vid 8q21.2, som till stor del har varit olösta under de senaste 20 åren,” skriver forskarna. Kluster av defensin spelar en nyckelroll i medfödd immunitet och strukturell variation i denna region har länge varit inblandad i mänsklig sjukdom.
den nya enheten, som adresserar alla fem av de tidigare otänkbara luckorna i det mänskliga referensgenomet, byggdes med en smart metod med flera datamängder, inklusive exakta långa läsningar: ”mer än hälften av PacBio HiFi-data finns i läser större än 17,8 kbp, med en mediannoggrannhet som överstiger 99,9%.”Efter ett ställningssteg baserat på Oxford Nanopore läser, contigs monterade från PacBio HiFi läser byttes in för att ge basparupplösningen. ”Vi förbättrade basparets noggrannhet i sekvensställningen genom att ersätta den råa ONT-sekvensen med flera konkordanta PacBio HiFi contigs”, rapporterar laget.
den kompletta chr8-sekvensen klockar in vid 146 Mb och innehåller mer än 3 Mb som saknas från GRCh38. Som Logsdon et al. skriv, ” resultatet är en helkromosomenhet med en uppskattad basparnoggrannhet som överstiger 99,99%.”
forskarna tacklade också det persnickety Bisexuell-defensin genkluster,” som vi löste in i en enda 7.06 Mbp—locus-väsentligt större än 4.56 Mbp-regionen i det nuvarande mänskliga referensgenomet, ” noterar de. Nästan alla sekvensdata-99.9934% av det, för att vara exakt — kom från HiFi läser. Den kompletta centromeren stod under tiden för 2, 08 Mb.
med denna vackra montering i handen tog T2T-teamet ut det för en snurrning. Först validerade de det med en mängd ortogonala verktyg, såsom optisk kartläggning. Därefter genererade de HiFi-data för kromosom 8-orthologerna i schimpans, makak och orangutan för att jämföra sekvensdata och rekonstruera den mänskliga autosomens evolutionära historia. ”Komparativa och fylogenetiska analyser visar att den högre ordningens satellitstruktur utvecklades specifikt i den stora apans förfader, och den centromeriska regionen utvecklades med en skiktad symmetri”, skriver laget. ”Vi uppskattar att mutationshastigheten för centromeriskt satellit-DNA accelereras minst 2.2-faldigt, och denna acceleration sträcker sig bortom den högre ordningens satelliten i den flankerande sekvensen.”
slutligen utförde forskarna en analys av fullängdsavskrifter som producerades med Iso-Seq-metoden. Den processen identifierade ”61 proteinkodande och 33 icke-kodande loci som kartlägger bättre till denna färdiga kromosom 8-sekvens än till GRCh38, inklusive upptäckten av nya gener som kartlägger för att kopiera antal polymorfa regioner”, rapporterar de. Tolv av dessa nya gener avslöjades i det knepiga bara för att det var en defensin locus.
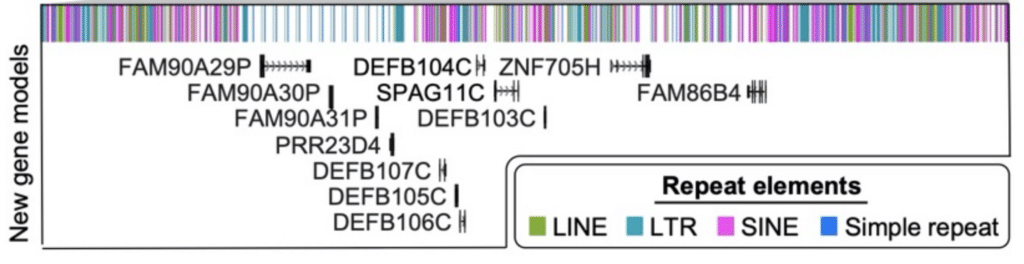
en kombination av HiFi – genommontering och RNA-annotering med Iso-Seq-data tillförde dessa 12 nya gener till regionen av kromosom 8 för defensin (defb). Logsdon, G et al. (2020)
för så många av oss i genomikgemenskapen representerar detta dokument mycket mer än sekvensen för en enda mänsklig kromosom. Det är ett uttalande om vad vetenskapen kan åstadkomma nu, och vart det kan leda oss under de kommande åren. Som författarna sammanfattade: ”Nu när komplexa regioner som dessa kan sekvenseras och monteras, kommer det att vara viktigt att utvidga dessa analyser till andra centromerer, flera individer och ytterligare arter för att förstå deras fulla inverkan med avseende på genetisk variation och evolution.”
du kan höra mer information från Logsdon direkt på en gratis onlinekonferens som är värd för T2T-konsortiet och Human Pangenome Reference Consortium (HPRC) den 22/23 September. Talare kommer att erbjuda nya insikter om kromosom 8 och rapportera om ytterligare T2T-framsteg mot en komplett mänsklig genommontering. Vid samma händelse kommer HPRC att presentera sin kompletterande insats för att sekvensera hundratals mänskliga genom till hög kvalitet. Bland presentatörerna finns: Karen Miga (@khmiga), Eric Green (@nhgri_director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@Mike_schatz), Heng li (@Lh3lh3) och många fler. Vi kommer att limmas på våra datorer för det och vi hoppas att du får chansen att gå med också!