betydelse
det finns ett växande intresse för circRNAs på grund av deras implikation i många biologiska processer och sjukdomar utöver deras biomarkörspotential. De upptäcks huvudsakligen av närvaron av läsningar som kartlägger deras backsplicing-korsning. Ändå är circRNAs inte längre de enda transkripten som innehåller en sådan korsning eftersom nya studier har visat att cirkulära DNA är vanliga och kan transkriberas vilket resulterar i transkript som skulle efterlikna en circRNA-signal. Därför kan denna nya typ av chimär transkript ändra hur circRNA-analys görs och påverka några av de resultat som redan rapporterats.
är cirkulära RNA de enda chimära transkripten?
cirkulärt RNA (circRNAs) återupptäcktes för några år sedan som icke-kanoniskt skarvade RNA-former närvarande i olika organismer inklusive människor (Salzman et al., 2012; Jeck et al., 2013). De är kovalent stängda transkript bildade genom en RNA-Back-splitsningshändelse, där en skarvdonator av en nedströms exon ansluter sig till en uppströms skarvacceptor som leder till kovalent stängda transkript som kännetecknas av närvaron av en back-splitsningskorsning som gör att circRNAs kan särskiljas från deras linjära motsvarigheter (Figur 1a) (Zhang et al., 2016; Wilusz, 2018).
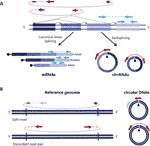
Figur 1 detektion av både cirkulärt RNA och DNA är baserat på läsningar som spänner över korsningen. (A) Backspliced sammanfogning mellan exoner och kanonisk exon sammanfogning avbildas med icke-kontinuerliga respektive kontinuerliga linjer som ger upphov till circRNAs och mRNAs. Parade ändläsningar som spänner över de backspliced korsningarna visas i rött och parade ändläsningar som överensstämmer med ett linjärt transkript visas i blått. (B) i likhet med circRNAs detekteras cirkulära DNA (eccDNAs) baserat på strukturella lästa varianter som överensstämmer med en cirkulariseringshändelse avbildad i rött .
sedan deras återupptäckt har det vetenskapliga samfundet uppmärksammat circRNAs och har undersökt deras engagemang i flera cellulära processer inom hälsa och sjukdom (Haque and Harries, 2017), deras potentiella roll som biomarkörer (Abu och Jamal, 2016) och deras regleringsfunktioner (Floris et al., 2016). CircRNAs är nu kända för att vara rikliga och stabila i cytosolen och kärnan (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015) och har också hittats fria i biofluider (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) och i extracellulära vesiklar (Kyoung Mi et al., 2017). Biomarkörspotentialen för circRNAs har studerats intensivt, i själva verket har det publicerats många fallkontrollstudier som söker differentiellt uttryckta circRNAs som kan vara biomarkörer för olika sjukdomar. Hittills har circRNAs varit inblandade i flera sjukdomar inklusive cancer (Kristensen et al., 2017; Arnaiz et al., 2018), neurologiska störningar (Akhter, 2018), hjärt-kärlsjukdomar (Aufiero et al., 2019) och immunrelaterade sjukdomar (Iparraguirre et al., 2017; Liu et al., 2019). På samma gång, att få till fullo förstå deras biogenes, egenskaper, funktioner, och konsekvenser i mänsklig biologi förblir som öppna frågor för forskare inom området.
även om funktionen hos de flesta circRNAs förblir okänd har det visat sig att vissa circRNAs kan fungera som microRNA-svampar, som reglerar microRNA-nivåerna och deras aktivitet (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). De är involverade i genuttrycksreglering genom att reglera transkriptionen av deras föräldragener, som konkurrerar med de linjära skarvnings-eller svampproteinerna (Ashwal-Fluss et al., 2014; Li et al., 2018). Intressant har ribosomprofileringsstudier nyligen visat att circRNAs också kan översättas både in vitro och in vivo (Legnini et al., 2017; Yang et al., 2017).
huvudfunktionen i circRNAs, och ansvarig för de flesta av deras speciella egenskaper, är deras cirkularitet. Därför, förutom att upptäcka deras karakteristiska back-spliced korsning, testa cirkulariteten hos dessa molekyler, är en av de viktigaste punkterna i varje circRNA-studie. Ändå har många studier baserat sin upptäckt av circRNAs på totalt RNA och kan därmed ha tolkat vissa linjära chimära transkript som circRNAs, vilket resulterar i falska positiva circRNA-detekteringar. För att kringgå detta problem har de flesta studier bekräftat cirkulariteten hos transkripten som hittats av total RNA-seq med RNase R, Northern blot eller electrophoretic methods (Jeck och Sharpless, 2014). Dessa cirkularitetsvalideringar har emellertid också ibland avslöjat transkript som verkar vara linjära snarare än cirkulära som bekräftar att detektering av circRNAs som börjar från totalt RNA kan leda till vissa falska positiva effekter. Dessa falska positiva effekter har tillskrivits tekniska artefakter eller transkript härledda från ovanliga händelser som exonduplikationer eller transplantationshändelser (Jeck och Sharpless, 2014; Szabo och Salzman, 2016). Med detta sagt har möjligheten att ha hittat sanna, biologiskt aktiva och funktionella linjära transkript som innehåller en sekvens som motsvarar en backsplicing-korsning (från och med nu kallad chimära linjära transkript) blivit något förbisedd eftersom en källa till sådant linjärt RNA inte har varit känt för friska celler.
cirkulära DNA som en källa till chimära linjära transkript
det mesta av det mänskliga genomet är organiserat i linjära kromosomer, men vissa undantag har länge accepterats såsom mitokondriellt DNA och kromosomavvikelser såsom DNA-cirklar som bär onkogener (t.ex. dubbla minuter) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017) och ringkromosomer (t bronkier et al., 2004). Det var inte förrän nyligen att olika cirkulära DNA: er som mikrodnas (Shibata et al., 2012) eller extrakromosomala cirkulära DNA (eccDNAs) befanns också uppstå från stora delar av olika eukaryota genom inklusive människa och jäst (m Obbller et al., 2015; Kumar et al., 2017; m Obbller et al., 2018).
cirkulära DNA bildas när två ändar av ett linjärt DNA är gemensamma tillsammans vilket resulterar i en korsning som liknar den backspliced korsningen på circRNAs som vanligtvis kallas brytpunktskorsning som detekteras baserat på strukturella lästa varianter som överensstämmer med en cirkulariseringshändelse (Gresham et al., 2010; m Obbller et al., 2018; Prada-Luengo et al., 2019) (Figur 1B). De sträcker sig vanligtvis från hundra baser till megabascirklar och kan innehålla fulla exoner och gener (Shibata et al ., 2012; m Obbller et al., 2015; Kumar et al., 2017; Turner et al., 2017; m Obbller et al., 2018) och medan vissa regioner i genomet oftare finns på cirkulärt DNA (Sinclair och Guarente, 1997;; m Obbller et al., 2016; Turner et al., 2017; m Obbller et al., 2018) verkar mest cirkulärt DNA förekomma slumpmässigt (Shibata et al., 2012; m Obbller et al., 2015; Kumar et al., 2017; m Obbller et al., 2018).
intressant, i ett nyligen publicerat papper m Obbller et al. identifierade tusentals eccDNAs i leukocyter och muskelceller i friska kontroller. Med tanken på att undersöka om eccDNAs kunde transkriberas, sekvenserades också ett mRNA-bibliotek från muskelvävnad och analyserades för transkriptionshändelser över brytpunktskorsningen av det detekterade eccDNA som hittade flera matchningar (m Occullar et al., 2018). Detta resultat tyder på att cirkulärt DNA i frisk vävnad transkriberas, vilket ger upphov till linjära och polyadenylerade transkript som kommer att bära en sekvens som motsvarar backsplicing-sekvensen av circRNAs (m Obbller et al., 2018) (Figur 2).
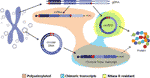
Figur 2 grafisk representation av olika transkript arised från antingen genomiskt DNA eller cirkulärt DNA. Exoner är färgade i lila och backspliced korsningar eller chimära korsningar visas i rött. Polyadenylerad, chimär korsning innehållande och RNAs r-resistenta transkript markeras i orange, blå respektive gul.
transkriptionsbeviset för cirkulära DNA, tillsammans med deras överflöd, leder oss till att föreslå att cirkulära DNA kan vara en naturlig källa till en väsentlig mängd linjära RNA som bär chimära korsningar. I många fall kan dessa chimära korsningar vara oskiljbara från backsplicing korsningar av circRNAs, och därför kan de vara förvirrande faktorer i circRNA-studier. I följande stycken kommer vi att förklara de uppgifter som stöder detta förslag.
circRNA upptäckt: Allt som glittrar är inte guld
som tidigare introducerats bildas circRNAs även om en icke-kanonisk skarvningshändelse kallas backsplicing. Transkript som härrör från denna backsplicing-händelse har en kovalent sluten slingstruktur med varken 5′-3′ polaritet eller en polyadenylerad svans och ännu viktigare kännetecknas de av närvaron av en krypterad exonordning i förhållande till det linjära transkriptet (Zhang et al., 2016; Wilusz, 2018). Denna krypterade exonordning blir tydlig i den backspliced korsningen som förbinder en 5′ nedströmssekvens med en uppströms 3′ – sekvens. Således utnyttjar alla circRNA-detekteringsalgoritmer närvaron av de bakre skarvade korsningarna som en diagnostisk egenskap för identifiering av circRNA (Figur 1a).
olika metoder har anpassats för detektering av dessa back-splitsade korsningar. Kommersiella matriser som innehåller sonder riktade mot dessa backspliced regioner har använts i stor utsträckning i biomarkörscreeningsstudier (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). Den efterföljande valideringen baseras ofta också på förstärkningen av backsplicing-korsningarna med hjälp av divergerande primers (Panda och Gorospe, 2018). Många andra artiklar har genomfört en hög genomströmning sekvensering analys som övervinner en av de viktigaste begränsningarna i arrayer gör det möjligt att upptäcka inte bara de kommenterade circRNAs men också de novo RNA cirkularisering händelser från genomiska regioner där ingen circRNA antecknades av tidigare studier. Flera bioinformatiska rörledningar har utvecklats för detektering av circRNAs i RNA-Seq-dataset, men alla är baserade på närvaron av läsningar som passerar över de bakre skarvkorsningarna och att hitta den mest tillförlitliga är fortfarande en utmaning för bioinformatiker (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
två huvudsakliga tillvägagångssätt kan följas för detektering av circRNAs i RNA-Seq-data. För det första är många circRNA RNA-Seq-studier baserade på RNAs r-behandlade prover för att tömma alla linjära rna före sekvensering. Även om detta tillvägagångssätt är speciellt utformat för circRNA-detektering är det värt att notera att RNAs r-nedbrytning är variabel, att det finns sällsynta fall av RNAs r-resistenta linjära RNA och RNAs r-känsliga circRNAs (Szabo och Salzman, 2016) och att denna behandling kräver en hög RNA-ingång som kan vara begränsande för vissa vävnader. Andra circRNA-studier väljer att sekvensera antingen totalt, ribosomalt utarmat (ribo -) eller icke-polyadenylerat (polyA-) RNA, där både linjära och cirkulära RNA kan hittas (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak et al., 2015). Detta tillvägagångssätt undviker användningen av RNAs R, vilket minskar RNA-mängden som behövs för sekvenseringen och gör det möjligt att studera uttrycket av andra typer av RNA från samma dataset. Det har visats att med ett bra sekvenseringsdjup och kvalitet och en noggrant dataanalys kan sanna circRNAs detekteras från total RNA-sekvensering (Wang et al., 2017), men i detta andra tillvägagångssätt behövs en senare cirkularitetsbekräftelse för circRNAs.
med upptäckten av linjära chimära rna transkriberade från cirkulära DNA är circRNAs inte längre de enda transkripten med chimära korsningar. Därför är det av yttersta vikt att notera att medan det första tillvägagångssättet kommer att väsentligt berika RNA-provet i circRNAs så att de flesta av de detekterade chimära korsningarna kommer att motsvara sanna circRNAs, kan den andra överskatta antalet circRNA-transkript genom att tillskriva circRNAs signalen som kommer från både circRNAs och de linjära chimära transkripten transkriberade från cirkulära DNA. Följaktligen, med hänsyn till samexistensen av circRNAs och linjära chimära transkript, behovet av cirkularitetstester och funktionalitetsanalyser får betydelse och särskild försiktighet bör iakttas när det gäller inte bara experimentella utan även beräkningsmetoder för att undvika att missa chimära transkript från cirkulära DNA med circRNAs bildade genom backsplicing.
diskussion
circRNA-fältet är fortfarande i ett tidigt skede, men circrna har redan visat sig vara förvånande molekyler, inblandade i många processer, med en stor biomarkörpotential och det kan också förändra hur vi förstår transkriptions-och översättningsprocesserna. Av dessa skäl får de uppmärksamhet och circRNA-fältet är för närvarande ett av de mest aktiva RNA-forskningsområdena. Det finns dock fortfarande många konflikter, kontroverser och öppna frågor (Li, 2019) som måste diskuteras.
i denna rapport, och mot bakgrund av de senaste framstegen inom det cirkulära DNA-fältet, vill vi påpeka transkriptionen från extrakromosomalt cirkulärt DNA som en av de viktigaste naturliga källorna till linjära transkript med back-splitsade signaler som kan störa circRNA-data (M Obbller et al., 2018). Från och med nu, förutom de tekniska artefakterna, dupliceringar och transplantationshändelser som kan leda till falska positiva effekter i circRNA-detekteringen, bör vi också ta hänsyn till förekomsten av denna nya typ av chimära transkript. Därför är cirkulariseringstester och funktionella analyser viktigare än någonsin.
i alla fall bör dessa chimära linjära transkript inte bara betraktas som en ren förvirrande faktor för circRNA-studier. Trots de tekniska implikationerna för circRNA-karakteriseringen, förekomsten av dessa circRNA-liknande chimära linjära RNA-molekyler som kommer från eccDNAs lägger till en ny typ av molekyl till den ständigt växande listan över RNA och utvidgar vår vision om transkriptomens komplexitet och dess reglering. Dessutom kan dessa linjära RNA-molekyler som kommer från eccDNA också presentera funktioner som liknar circRNA, inklusive regulatoriska funktioner eller potentialen att översättas. Genprodukter från eccDNA-transkript kan potentiellt bidra till fenotypen av somatiska celler och vävnad som rapporterats i jäst (Gresham et al., 2010; Demeke et al., 2015). Men i detta växande fält behövs mer data och forskning för att börja skrapa ytan på isberget.
Författarbidrag
LI, IP-L, BR Och do skrev papperet.
finansiering
denna studie har finansierats av Instituto de Salud Carlos III genom projektet ”PI17/00189” (samfinansierat av Europeiska regionala utvecklingsfonden/Europeiska socialfonden) ”investera i din framtid”). IP-L och BR stöddes av danska rådet för oberoende forskning, 6108 – 00171b och LI stöddes av utbildningsdepartementet i den baskiska regeringen .
intressekonflikt
författarna förklarar att forskningen genomfördes i avsaknad av kommersiella eller finansiella relationer som kan tolkas som en potentiell intressekonflikt.