luni, 21 septembrie 2020
chiar și în domeniul genomicii, unde apar noi descoperiri la fiecare câteva luni, finalizarea primului Autozom uman complet secvențiat este o realizare importantă. Foarte precis, fără lacune, fără îmbinări greșite-doar cromozomul 8 în toată gloria sa. Este un feat remarcabil și suntem onorați că PacBio HiFi reads a jucat un rol esențial în a ajuta la realizarea acestuia.
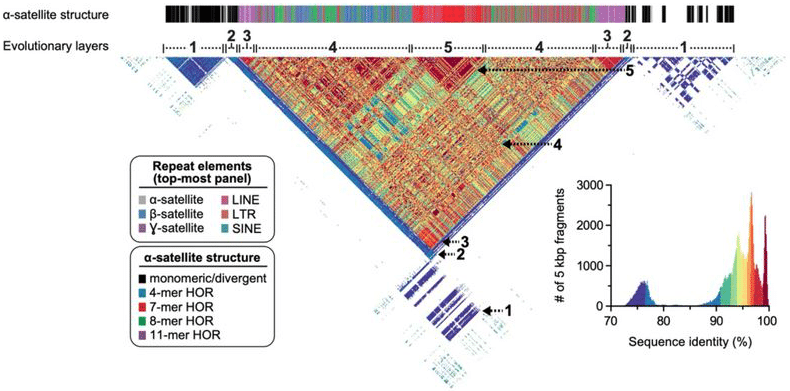
secvența completă de centromere a cromozomului 8 arată o diversitate de repetări prin satelit și alte repetări genomice abundente, acum cu o rezoluție aproape perfectă la nivel de bază de la capăt la capăt. Logsdon, G și colab. (2020)
această lucrare este descrisă într-o preimprimare postată recent la bioRxiv de la autorul principal Glennis Logsdon (@glennis_logsdon), autorul principal Evan Eichler și colaboratorii lor din consorțiul telomeri-la-telomeri (T2T). Face parte din inițiativa mai largă T2T de a secventa și asambla primul genom uman cu adevărat complet și urmează eliberarea anterioară a cromozomului X complet secvențiat.
„de la anunțarea secvențierii genomului uman în urmă cu 20 de ani, cromozomii umani au rămas neterminați din cauza regiunilor mari de repetări foarte identice situate în centromere, duplicarea segmentară și brațele scurte acrocentrice ale cromozomilor”, notează autorii. „Apariția tehnologiilor de secvențiere citite de mult timp și a algoritmilor asociați au făcut acum posibilă asamblarea sistematică a acestor regiuni din ADN-ul nativ pentru prima dată.”
cromozomul 8 a făcut o țintă atractivă pentru primul Autozom al T2T datorită centromerului său gestionabil (estimat anterior la 1,5 Mb până la 2,2 Mb lungime). Dar cromozomul găzduiește ,de asemenea, ” una dintre cele mai dinamice regiuni din punct de vedere structural din genomul uman—clusterul genic al genei-defensin situat la 8p23.1—precum și un neocentromer situat la 8q21.2, care au fost în mare parte nerezolvate în ultimii 20 de ani”, scriu oamenii de știință. Β-defensin grup joacă un rol esențial în imunitatea înnăscută și structurale variație în această regiune a fost mult timp implicat în boli umane.
noul ansamblu, care abordează toate cele cinci lacune care nu au putut fi rezolvate anterior în genomul de referință uman, a fost construit cu o metodă inteligentă folosind mai multe seturi de date, inclusiv citiri lungi precise: „mai mult de jumătate din datele PacBio HiFi sunt conținute în citiri mai mari de 17,8 kbp, cu o precizie mediană care depășește 99,9%.”După o etapă de schelă bazată pe Oxford Nanopore reads, contigs asamblate de la pacbio HiFi reads au fost schimbate pentru a oferi rezoluția perechii de bază. „Am îmbunătățit precizia perechii de bază a schelelor secvenței prin înlocuirea secvenței ont brute cu mai multe contig-uri concordante PacBio HiFi”, relatează echipa.
secvența chr8 completă se fixează la 146 Mb și include mai mult de 3 Mb lipsă din GRCh38. As Logsdon și colab. scrieți: „rezultatul este un ansamblu de cromozomi întregi cu o precizie estimată a perechii de baze care depășește 99,99%.”
oamenii de știință au abordat, de asemenea, acel grup de gene persnickety-defensin, „pe care l—am rezolvat într-un singur locus de 7,06 Mbp-substanțial mai mare decât regiunea de 4,56 Mbp din genomul de referință uman actual”, notează ei. Aproape toate aceste date de secvență — 99,9934% din acestea, pentru a fi precise — provin din citirile HiFi. Între timp, centromerul complet a reprezentat 2, 08 Mb.
cu acest ansamblu frumos în mână, echipa T2T a scos-o pentru o rotire. În primul rând, l-au validat cu o serie de instrumente ortogonale, cum ar fi cartografierea optică. Apoi, au generat date HiFi pentru cromozomul 8 ortologi în cimpanzeu, macac, și orangutan pentru a compara datele secvenței și a reconstrui istoria evolutivă a autozomului uman. „Analizele comparative și filogenetice arată că structura satelitului de ordinul superior al satelitului a evoluat în mod specific în strămoșul maimuței mari, iar regiunea centromerică a evoluat cu o simetrie stratificată”, scrie echipa. „Estimăm că rata de mutație a ADN-ului prin satelit centromeric este accelerată cel puțin 2.De 2 ori, iar această accelerație se extinde dincolo de satelitul de ordin superior de ordinul X-X în secvența de flancare.”
în cele din urmă, cercetătorii au efectuat o analiză a transcrierilor complete produse cu metoda Iso-Seq. Acest proces a identificat „61 de codificare a proteinelor și 33 de loci necodificatori care se mapează mai bine la această secvență de cromozom 8 terminată decât la GRCh38, inclusiv descoperirea unor noi cartografieri de gene pentru a copia regiunile polimorfe ale numărului”, raportează ei. Douăsprezece dintre aceste noi gene au fost descoperite doar în acel Locus complicat de apărare a sistemului de apărare.
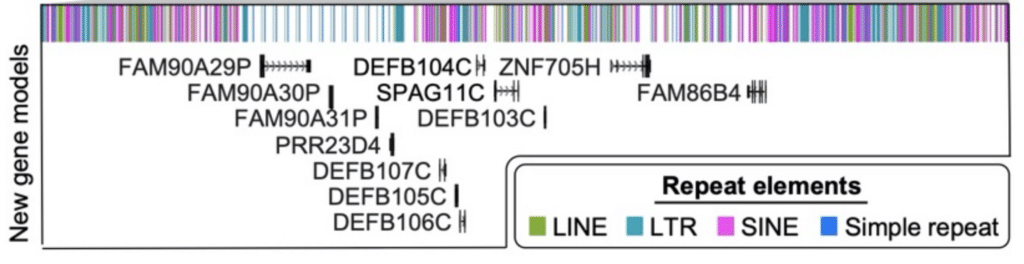
o combinație de asamblare a genomului HiFi și adnotare ARN cu date Iso-Seq a adăugat aceste 12 gene noi în regiunea de apărare a cromozomului 8 (defb). Logsdon, G și colab. (2020)
pentru atât de mulți dintre noi din comunitatea genomică, această lucrare reprezintă mult mai mult decât secvența unui singur cromozom uman. Este o declarație despre ceea ce știința poate realiza acum și unde ne poate duce asta în anii următori. După cum au rezumat autorii: „Acum că regiuni complexe precum acestea pot fi secvențiate și asamblate, va fi important să extindem aceste analize la alți centromeri, mai mulți indivizi și specii suplimentare pentru a înțelege impactul lor deplin în ceea ce privește variația și evoluția genetică.”
puteți auzi mai multe detalii de la Logsdon direct la o conferință online gratuită găzduită de Consorțiul T2T și consorțiul de referință Pangenome uman (HPRC) pe 22/23 septembrie. Vorbitorii vor oferi noi informații despre cromozomul 8 și vor raporta progresele ulterioare ale T2T către o asamblare completă a genomului uman. În cadrul aceluiași eveniment, HPRC își va prezenta efortul complementar de a secventa sute de genomi umani la o calitate înaltă. Prezentatorii includ: Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng li (@Lh3lh3) și multe altele. Vom fi lipite la computerele noastre pentru ea și sperăm că veți avea o șansă să se alăture, de asemenea!