poniedziałek, 21 września 2020
nawet w dziedzinie genomiki, gdzie co kilka miesięcy pojawiają się nowe przełomy, Ukończenie pierwszego w pełni zsekwencjonowanego ludzkiego autosomu jest doniosłym osiągnięciem. Bardzo dokładne, bez luk, bez błędnych połączeń – tylko chromosom 8 w całej okazałości. To niezwykły wyczyn i jesteśmy zaszczyceni, że PacBio HiFi reads odegrało kluczową rolę w jego osiągnięciu.
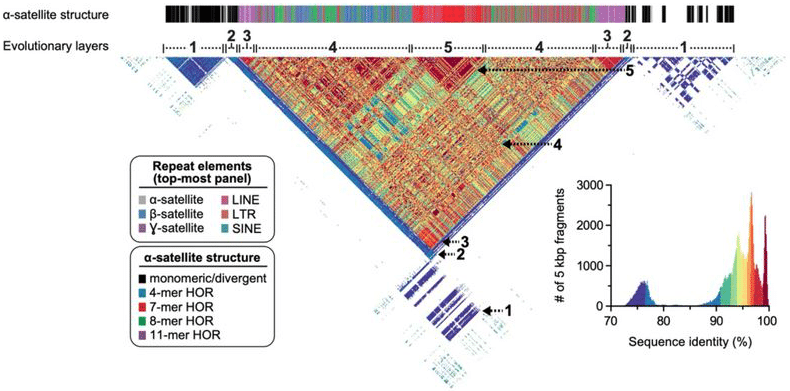
kompletna Sekwencja centromerowa chromosomu 8 pokazuje różnorodność powtórzeń satelitarnych i innych obfitych powtórzeń genomowych, teraz z niemal idealną rozdzielczością na poziomie podstawowym od końca do końca. Logsdon, G et al. (2020)
praca ta została opisana w preprincie opublikowanym niedawno w bioRxiv przez główną autorkę Glennis Logsdon (@glennis_logsdon), starszego autora Evana Eichlera i ich współpracowników w Konsorcjum Telomere-to-Telomere (T2T). Jest to część szerszej inicjatywy T2T mającej na celu zsekwencjonowanie i zmontowanie pierwszego prawdziwie kompletnego ludzkiego genomu i następuje po wcześniejszym uwolnieniu w pełni zsekwencjonowanego chromosomu X.
„od ogłoszenia sekwencjonowania ludzkiego genomu 20 lat temu, ludzkie chromosomy pozostały niedokończone z powodu dużych regionów wysoce identycznych powtórzeń znajdujących się w centromerach, segmentowego duplikacji i akcentrycznych krótkich ramion chromosomów”, autorzy zauważają. „Pojawienie się technologii sekwencjonowania z długim odczytem i powiązanych algorytmów umożliwiło po raz pierwszy systematyczne łączenie tych regionów z rodzimego DNA.”
chromosom 8 był atrakcyjnym celem dla pierwszego autosomu T2T ze względu na kontrolowany centromer (wcześniej szacowany na 1,5 Mb do 2,2 Mb długości). Ale chromosom jest również domem dla ” jednego z najbardziej dynamicznych strukturalnie regionów ludzkiego genomu—klastra genów β-defenzyny zlokalizowanego w 8p23.1—a także neocentromeru zlokalizowanego w 8q21.2, które były w dużej mierze nierozwiązane przez ostatnie 20 lat ” – piszą naukowcy. Klaster β-defenzyny odgrywa kluczową rolę w odporności wrodzonej, a zmiany strukturalne w tym regionie od dawna są związane z chorobami człowieka.
nowy zespół, który rozwiązuje wszystkie pięć wcześniej trudnych do usunięcia luk w ludzkim genomie referencyjnym, został zbudowany za pomocą sprytnej metody wykorzystującej kilka zestawów danych, w tym dokładne długie odczyty: „ponad połowa danych PacBio HiFi zawiera odczyty większe niż 17,8 kbp, z medianą dokładności przekraczającą 99,9%.”Po etapie rusztowania opartym na Oxford Nanopore reads, contigs zmontowane z PacBio HiFi reads zostały zamienione, aby zapewnić rozdzielczość pary bazowej. „Poprawiliśmy dokładność par bazowych rusztowań sekwencyjnych, zastępując surową sekwencję ONT kilkoma zgodnymi ciągami PacBio HiFi”, donosi zespół.
kompletna Sekwencja chr8 zegara na 146 Mb i zawiera więcej niż 3 Mb brakuje w GRCh38. Jako Logsdon et al. napisz: „rezultatem jest zespół całego chromosomu z szacowaną dokładnością pary zasad przekraczającą 99,99%.”
naukowcy zajęli się również tym klastrem genów persnickety β-defenzyny, „który rozpadł się na pojedyncze locus 7,06 Mbp—znacznie większe niż region 4,56 MBP w obecnym ludzkim genomie referencyjnym”, zauważają. Prawie wszystkie dane o sekwencji — a dokładniej 99,9934% – pochodziły z odczytów HiFi. Natomiast kompletny centromer liczył 2,08 Mb.
z tym pięknym montażem w ręku, zespół T2T wziął go na przejażdżkę. Po pierwsze, walidowali go za pomocą wielu narzędzi ortogonalnych, takich jak mapowanie optyczne. Następnie wygenerowali dane HiFi Dla ortologów chromosomu 8 U szympansa, makaka i orangutana, aby porównać dane sekwencji i zrekonstruować ewolucyjną historię ludzkiego autosomu. „Analizy porównawcze i filogenetyczne pokazują, że struktura α-satelitów wyższego rzędu ewoluowała szczególnie u przodka Wielkiej małpy, a region centromeryczny ewoluował z warstwową symetrią”, pisze zespół. „Szacujemy, że szybkość mutacji centromerycznego DNA satelitarnego jest przyspieszana co najmniej 2.2-krotnie, a przyspieszenie to rozciąga się poza satelitę wyższego rzędu α w sekwencję flankującą.”
wreszcie naukowcy przeprowadzili analizę pełnowymiarowych transkryptów wytworzonych metodą Iso-Seq. Proces ten zidentyfikował „61 kodujących białko i 33 niekodujące loci, które mapują lepiej do tej gotowej sekwencji chromosomu 8 niż do GRCh38, w tym odkrycie nowych genów mapujących do kopiowania liczb polimorficznych regionów”, raportują. Dwanaście z tych nowych genów zostało odkrytych w samym locus β-defenzyny.
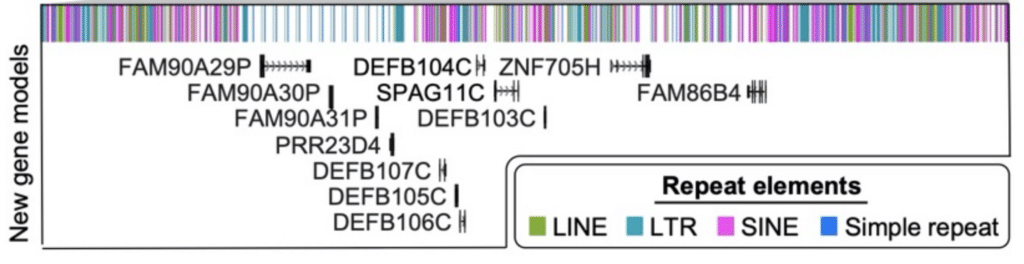
połączenie montażu genomu HiFi i adnotacji RNA z danymi Iso-Seq dodało te 12 nowych genów do regionu β-defenzyny (DEFB) chromosomu 8. Logsdon, G et al. (2020)
dla tak wielu z nas w społeczności genomicznej, ten artykuł reprezentuje znacznie więcej niż sekwencję pojedynczego ludzkiego chromosomu. To Oświadczenie o tym, co nauka może teraz osiągnąć i dokąd nas to zaprowadzi w nadchodzących latach. Jak podsumowali autorzy: „Teraz, gdy złożone regiony, takie jak te, mogą być sekwencjonowane i montowane, ważne będzie rozszerzenie tych analiz na inne centromery, wiele osobników i dodatkowe gatunki, aby zrozumieć ich pełny wpływ w odniesieniu do zmienności genetycznej i ewolucji.”
więcej informacji od Logsdon można usłyszeć bezpośrednio na bezpłatnej konferencji online współorganizowanej przez konsorcjum T2T i Human Pangenome Reference Consortium (HPRC) w dniach 22/23 września. Prelegenci przedstawią nowe spostrzeżenia na temat chromosomu 8 i raport na temat dalszych postępów T2T w kierunku kompletnego montażu ludzkiego genomu. Podczas tego samego wydarzenia, HPRC zaprezentuje swój komplementarny wysiłek w celu sekwencjonowania setek ludzkich genomów do wysokiej jakości. Prezenterzy: Karen Miga (@khmiga), Eric Green (@nhgri_director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@Mike_schatz), Heng Li (@Lh3lh3) i wielu innych. Będziemy przyklejeni do naszych komputerów za to i mamy nadzieję, że będziesz miał szansę dołączyć, jak również!