Significance
istnieje rosnące zainteresowanie cirrna ze względu na ich wpływ na wiele procesów biologicznych i chorób, oprócz ich potencjału biomarkerowego. Są one wykrywane głównie przez obecność odczytów mapujących ich złącze backsplicing. Niemniej jednak, cirrna nie są już jedynymi transkryptami zawierającymi takie połączenie, ponieważ ostatnie badania wykazały, że koliste DNA są powszechne i mogą być transkrybowane, co skutkuje transkrypcjami, które naśladowałyby sygnał cirrna. Dlatego ten nowy typ chimerycznej transkrypcji może zmienić sposób, w jaki przeprowadzana jest analiza cirrna i wpłynąć na niektóre z już zgłoszonych wyników.
czy okrągłe RNA to jedyne chimeryczne transkrypcje?
okrągłe RNA (cirrnas) zostały odkryte kilka lat temu jako niekanonicznie splecione formy RNA obecne w różnych organizmach, w tym u ludzi (Salzman et al., 2012; Jeck et al., 2013). Są one kowalencyjnie zamkniętymi transkryptami powstałymi w wyniku splicingu wstecznego RNA, w którym dawca splicingu z niższego egzonu przyłącza się do akceptora splicingu wyższego, co prowadzi do kowalencyjnie zamkniętych transkryptów, które charakteryzują się obecnością połączenia splicingu wstecznego, co sprawia, że cirrna są odróżnialne od ich liniowych odpowiedników (Fig., 2016; Wilusz, 2018).
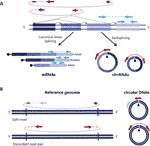
Fig. 1 wykrywanie zarówno kolistego RNA, jak i DNAs opiera się na odczytach rozciągających się na złączu. A) wsteczne łączenie między eksonami i kanoniczne łączenie eksonów są przedstawione za pomocą linii nieciągłych i ciągłych, odpowiednio, dając początek cirrna i mRNA. Sparowane odczyty końców rozciągające się na tylnych złączach są pokazane na czerwono, a sparowane odczyty końców zgodne z transkrypcją liniową są pokazane na niebiesko. (B) podobne do cirrna, okrągłe DNAs (eccdna) są wykrywane na podstawie wariantów strukturalnych odczytu zgodnych z wydarzeniem circularization przedstawionym na Czerwono .
od czasu ich ponownego odkrycia społeczność naukowa zwróciła uwagę na cirrna i zbadała ich udział w kilku procesach komórkowych w zdrowiu i chorobie (Haque and Harries, 2017), ich potencjalną rolę jako biomarkerów (Abu and Jamal, 2016) oraz ich funkcje regulacyjne (Floris et al., 2016). Cirrnas są obecnie znane jako obfite i stabilne w cytozolu i jądrze (Salzman et al., 2012; Jeck et al., 2013; Li et al., 2015), a także stwierdzono wolne w biofluidach (Bahn et al., 2015; Memczak et al., 2015; Chen et al., 2018) oraz w pęcherzykach pozakomórkowych (Kyoung mi et al., 2017). Potencjał biomarkerów cirrna był intensywnie badany, w rzeczywistości opublikowano wiele badań kontrolnych dotyczących przypadków szukających różnicowo wyrażonych cirrna, które mogą być biomarkerami różnych chorób. Do tej pory cirrnas były zaangażowane w kilka chorób, w tym raka (Kristensen et al., 2017; Arnaiz et al., 2018), zaburzenia neurologiczne (Akhter, 2018), choroby układu krążenia (Aufiero et al., 2019) i chorób immunologicznych (Iparraguirre et al., 2017; Liu et al., 2019). Jednocześnie pełne zrozumienie ich biogenezy, cech, funkcji i implikacji w biologii człowieka pozostaje kwestią otwartą dla badaczy w tej dziedzinie.
chociaż funkcja większości cirrna pozostaje nieznana, wykazano, że niektóre cirrna mogą działać jako gąbki mikroRNA, regulując poziomy mikroRNA i ich aktywność (Hansen et al., 2013; Memczak et al., 2013; Zheng et al., 2016). Biorą udział w regulacji ekspresji genów poprzez regulację transkrypcji ich genów rodzicielskich, konkurując z liniowym splicingiem lub białkami gąbczastymi(Ashwal-Fluss et al., 2014; Li et al., 2018). Co ciekawe, badania profilowania rybosomów wykazały ostatnio, że cirrnas można również przetłumaczyć zarówno in vitro, jak i In vivo (Legnini et al., 2017; Yang et al., 2017).
główną cechą cirrna i odpowiedzialną za większość ich specjalnych właściwości jest ich kolistość. Dlatego, oprócz wykrycia ich charakterystycznego połączenia z tyłu, badanie kolistości tych cząsteczek jest jednym z kluczowych punktów w każdym badaniu cirrna. Niemniej jednak, wiele badań oparło swoje odkrycie cirrna na całkowitym RNA i mogło w ten sposób zinterpretować niektóre liniowe transkrypty chimeryczne jako cirrna, co skutkowało fałszywie dodatnimi wykrywaniami cirrna. Aby obejść ten problem, większość badań potwierdziło kolistość transkryptów znalezionych przez całkowite RNA-seq przy użyciu RNazy R, Northern blot lub metod elektroforetycznych (Jeck and Sharpless, 2014). Jednakże te walidacje kolistości ujawniły czasami transkrypty, które wydają się być liniowe, a nie kołowe, potwierdzające, że wykrywanie cirrna zaczynających się od całkowitego RNA może prowadzić do fałszywych alarmów. Te fałszywe alarmy zostały przypisane technicznym artefaktom lub transkryptom pochodzącym z niezwykłych zdarzeń, takich jak duplikacje egzonów lub przeszczepy (Jeck and Sharpless, 2014; Szabo and Salzman, 2016). To powiedziawszy, opcja znalezienia prawdziwych, biologicznie aktywnych i funkcjonalnych transkryptów liniowych, które zawierają sekwencję odpowiadającą złączu backsplicing (od teraz nazywanym chimerycznymi transkryptami liniowymi), została nieco pominięta, ponieważ źródło takiego liniowego RNA nie było znane dla zdrowych komórek.
koliste DNA jako źródło chimerycznych transkryptów liniowych
Większość ludzkiego genomu jest zorganizowana w liniowe chromosomy, jednak od dawna akceptowane są pewne wyjątki, takie jak mitochondrialne DNA i aberracje chromosomowe, takie jak kręgi DNA niosące onkogeny (np. podwójne minuty) (Benner et al., 1991; Nathanson et al., 2014; Turner et al., 2017)i chromosomy pierścieniowe (Tümer et al., 2004). Dopiero niedawno różne okrągłe DNA, takie jak mikrodna (Shibata et al., 2012) lub pozachromosomalne okrągłe DNA (eccDNAs) również powstały z dużej części różnych genomów eukariotycznych, w tym ludzkich i drożdży (Møller et al., 2015; Kumar et al., 2017; Møller et al., 2018).
okrągłe DNAs powstają, gdy dwa końce liniowego DNA są połączone ze sobą, co daje połączenie podobne do złącza backsplicated na cirrna powszechnie zwanego złączem punktu przerwania, które jest wykrywane w oparciu o warianty odczytu strukturalnego zgodne z zdarzeniem cyrkulacyjnym (Gresham et al., 2010; Møller et al., 2018; Prada-Luengo et al., 2019) (rys. 1B). Zazwyczaj wahają się od stu podstaw do мегабазных okrążeń i mogą zawierać pełne экзоны i geny (Шибата i in., 2012; Möller i wsp., 2015; Kumar i wsp., 2017; Turner i in., 2017; Möller i wsp., 2018), i chociaż niektóre obszary genomu częściej spotykane w rondzie DNA (Sinclair i Гуаренте, 1997;; Möller i wsp., 2016; Turner i in., 2017; Möller i wsp., 2018), większość pierścieniowych DNA, wydaje się, występują losowo ( Шибата i in., 2012; Möller i wsp., 2015; Kumar i wsp., 2017; Meller, itp.., 2018).
co ciekawe, w ostatnim artykule Moeller i wsp. zidentyfikowano tysiące eccdna w leukocytach i komórkach mięśniowych w zdrowych kontrolach. Z pomysłem zbadania, czy eccdna można transkrybować, biblioteka mRNA została również zsekwencjonowana z tkanki mięśniowej i przeanalizowana pod kątem zdarzeń transkrypcyjnych przez skrzyżowanie punktu przerwania wykrytego eccDNA znajdując kilka dopasowań(Møller et al., 2018). To odkrycie sugeruje, że koliste DNA w zdrowej tkance jest transkrybowane, dając początek liniowym i poliadenylowanym transkryptom, który będzie zawierał sekwencję równoważną sekwencji backsplicingu cirrna(Møller et al., 2018) (Rys. 2).
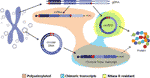
Rysunek 2 Graficzna reprezentacja różnych transkryptów powstających z dna genomowego lub DNA okrągłego. Egzony są zabarwione na fioletowo, a tylne złącza lub chimeryczne są pokazane na Czerwono. Poliadenylowane, chimeryczne złącze zawierające i RNase r oporne transkrypty są podświetlone odpowiednio w kolorze pomarańczowym, niebieskim i żółtym.
transkrypcyjne dowody kolistych DNAs, wraz z ich obfitością, prowadzą nas do sugerowania, że koliste DNAs mogą być naturalnym źródłem znacznej ilości liniowych RNA niosących połączenia chimeryczne. W wielu przypadkach te połączenia chimeryczne mogą być nie do odróżnienia od połączeń wstecznych cirrna, a zatem mogą być czynnikami zakłócającymi w badaniach cirrna. W kolejnych akapitach wyjaśnimy dane wspierające ten wniosek.
wykrywanie cirrna: Wszystko, co świeci, nie jest złotem
jak wcześniej wprowadzono, cirrna powstają poprzez niekanoniczne splicing zwane backsplicing. Transkrypty wynikające z tego zdarzenia wstecznego mają kowalencyjnie zamkniętą strukturę pętli bez polaryzacji 5 ’-3′, ani poliadenylowanego ogona, a co ważniejsze, charakteryzują się obecnością kodowanego porządku eksonu w stosunku do transkrypcji liniowej (Zhang et al., 2016; Wilusz, 2018). Ten zakodowany porządek eksonu staje się widoczny w złączu backspliced, który łączy sekwencję 5 'downstream z sekwencją 3′ upstream. Tak więc wszystkie algorytmy detekcji cirrna wykorzystują obecność tylnych połączeń jako cechę diagnostyczną do identyfikacji cirrna (rysunek 1a).
różne metody zostały przystosowane do wykrywania tych tylnych połączeń. Komercyjne tablice zawierające sondy ukierunkowane na te backsplicated regionów były szeroko stosowane w badaniach przesiewowych biomarkerów (Iparraguirre et al., 2017; Liu et al., 2017; Sui et al., 2017; Li et al., 2018). Późniejsza Walidacja jest często również oparta na wzmocnieniu połączeń backsplicingu przy użyciu rozbieżnych starterów (Panda i Gorospe, 2018). Wiele innych prac przeprowadziło analizę sekwencjonowania o wysokiej przepustowości, która pokonuje jedno z głównych ograniczeń tablic, pozwalając wykryć nie tylko adnotowane cirrna, ale także De novo zdarzenia cyrkularyzacji RNA z regionów genomowych, w których żadne cirrna nie były adnotowane w poprzednich badaniach. Kilka bioinformatycznych rurociągów zostało opracowanych do wykrywania cirrna w zestawach danych RNA-Seq, ale wszystkie z nich opierają się na obecności reads crossing over back-splicing junctions i znalezienie najbardziej wiarygodnego jest nadal wyzwaniem dla bioinformatyków (Hansen et al., 2016; Hansen, 2018; Prada-Luengo et al., 2019).
można zastosować dwa główne podejścia do wykrywania cirrna w danych RNA-Seq. Po pierwsze, wiele badań cirrna RNA-seq opiera się na próbkach poddanych działaniu RNazy R w celu wyczerpania wszystkich liniowych RNA przed sekwencjonowaniem. Chociaż to podejście jest specjalnie zaprojektowane do wykrywania cirrna, warto zauważyć, że degradacja RNazy R jest zmienna, że istnieją rzadkie przypadki liniowych RNA opornych na rnazę R i wrażliwych na rnazę r cirrna (Szabo i Salzman, 2016) i że leczenie to wymaga wysokiego wejścia RNA, które może ograniczać niektóre tkanki. Inne badania cirrna wybierają sekwencję całkowitego, zubożonego rybosomalnie (rybo -) lub nie-poliadenylowanego (polyA -) RNA, gdzie można znaleźć zarówno liniowe, jak i kołowe RNA (Salzman et al., 2012; Memczak et al., 2013; Broadbent et al., 2015; Lu et al., 2015; Memczak i in., 2015). Takie podejście pozwala uniknąć użycia RNazy R, która zmniejsza ilość RNA potrzebną do sekwencjonowania i pozwala na badanie ekspresji innych typów RNA z tego samego zbioru danych. Wykazano, że przy dobrej głębokości i jakości sekwencjonowania oraz starannej analizie danych, prawdziwe cirrna można wykryć z całkowitego sekwencjonowania RNA (Wang et al., 2017), jednak w tym drugim podejściu potrzebne jest późniejsze potwierdzenie circrna.
wraz z odkryciem liniowych chimerycznych RNA transkrybowanych z okrągłych DNAs, cirrna nie są już jedynymi transkryptami z chimerycznymi złączami. Dlatego niezwykle ważne jest, aby zauważyć, że podczas gdy pierwsze podejście znacząco wzbogaci próbkę RNA w cirrna tak, że większość wykrytych połączeń chimerycznych będzie odpowiadać prawdziwym cirrna, drugie może przecenić liczbę transkryptów cirrna, przypisując cirrna sygnał pochodzący zarówno z cirrna, jak i liniowych transkryptów chimerycznych transkrybowanych z okrągłych DNAs. W związku z tym, biorąc pod uwagę współistnienie cirrna i liniowych transkryptów chimerycznych, potrzeba testów kołowości i testów funkcjonalności zyskuje na znaczeniu i należy zachować szczególną ostrożność nie tylko w odniesieniu do metod eksperymentalnych, ale także obliczeniowych, aby uniknąć mylenia chimerycznych transkryptów z okrągłych DNA z cirrna utworzonymi przez backsplicing.
dyskusja
pole cirrna jest jeszcze na wczesnym etapie, jednak cirrna już okazały się zdumiewającymi cząsteczkami, zaangażowanymi w wiele procesów, o wielkim potencjale biomarkera, co może również zmienić sposób, w jaki rozumiemy procesy transkrypcji i translacji. Z tych powodów przyciągają uwagę, a pole cirrna jest obecnie jedną z najbardziej aktywnych dziedzin badań RNA. Wciąż jednak istnieje wiele konfliktów, kontrowersji i otwartych pytań (Li, 2019), które należy omówić.
w tym raporcie, w świetle ostatnich postępów w dziedzinie okrągłego DNA, chcemy zwrócić uwagę na transkrypcję z pozachromosomalnego okrągłego DNA jako jedno z głównych naturalnych źródeł liniowych transkryptów z wstecznie splecionymi sygnałami, które mogą zakłócać dane cirrna (Møller et al., 2018). Od teraz, oprócz technicznych artefaktów, duplikacji i przeszczepiania zdarzeń, które mogą prowadzić do fałszywych alarmów w wykrywaniu cirrna, powinniśmy również wziąć pod uwagę istnienie tego nowego rodzaju transkryptów chimerycznych. Dlatego testy cyrkulacyjne i testy funkcjonalne są ważniejsze niż kiedykolwiek.
w każdym razie te chimeryczne transkrypty liniowe powinny być uważane nie tylko za zwykły czynnik zakłócający badania cirrna. Pomimo technicznych implikacji dla charakterystyki cirrna, istnienie tych podobnych do cirrna chimerycznych liniowych cząsteczek RNA pochodzących z eccdna dodaje nowy typ cząsteczki do stale rosnącej listy RNA i rozszerza naszą wizję złożoności transkryptomu i jego regulacji. Co więcej, te liniowe cząsteczki RNA pochodzące z eccDNA mogą również prezentować funkcje podobne do cirrna, w tym funkcje regulacyjne lub potencjał do translacji. Produkty genowe z transkryptów eccDNA mogą potencjalnie przyczynić się do fenotypu komórek i tkanek somatycznych, jak opisano u drożdży (Gresham et al., 2010; Demeke et al., 2015). Jednak w tej rodzącej się dziedzinie potrzebne są więcej danych i badań, aby zacząć drapać powierzchnię góry lodowej.
LI, IP-L, BR I DO napisał artykuł.
finansowanie
to badanie zostało sfinansowane przez Instituto de Salud Carlos III w ramach projektu „PI17/00189” (współfinansowanego ze środków Europejskiego Funduszu Rozwoju Regionalnego/Europejskiego Funduszu Społecznego) „inwestowanie w Twoją przyszłość”). IP-L I BR były wspierane przez duńską Radę niezależnych badań naukowych, 6108-00171B, A LI był wspierany przez Departament Edukacji rządu baskijskiego .
konflikt interesów
autorzy oświadczają, że badania zostały przeprowadzone przy braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.