- Introduksjon
- Metoder
- Filtrering Av Ribosomale Profileringsdata
- Montering Av Virtuelle Genomer
- Bestemmelse Av Ribo-seq-Lesekartet Region på ET Veikryss (RMRJ) av circRNAs
- Opplæring Av Modell Og Klassifisering Av RMRJs
- Prediksjon Av Oversatte Peptider Av RMRJs
- Resultater Og Diskusjon
- Oversatt circRNAs I Mennesker Og A. thaliana
- Funksjonell Berikelse Av Menneske og a. thaliana circRNAs Med Kodingspotensial
- Nøyaktighetstest For Sirkkode
- Påvirkning Av Ribo-seq Datasekvenseringsdybde
- Sammenligning Av Kretskode Med Andre Verktøy
- Konklusjoner
- Tilgjengelighet Og Krav
- Datatilgjengelighetserklæring
- Forfatterbidrag
- Finansiering
- Interessekonflikter
- Supplerende Materiale
Introduksjon
Sirkulære Rna (circrna) er en spesiell type ikke-kodende RNA-molekyl som har blitt et hett forskningsemne innen RNA og får stor oppmerksomhet (Chen Og Yang, 2015). Sammenlignet med tradisjonelle lineære Rna (som inneholder 5′ og 3′ ender), har circRNA-molekyler vanligvis en lukket sirkulær struktur; gjør dem mer stabile og mindre utsatt for nedbrytning (Vicens and Westhof, 2014). Selv om eksistensen av circrna har vært kjent i noen tid, ble disse molekylene ansett som et biprodukt AV RNA-spleising. Men med utviklingen av høy gjennomstrømning sekvensering og bioinformatikk teknologier, circRNAs har blitt anerkjent i dyr og planter (Chen Og Yang, 2015). Nyere studier har også vist at et stort antall circrna kan oversettes til små peptider i celler (Pamudurti et al., 2017) og har sentrale roller til tross for deres noen ganger lave uttrykksnivå (Hsu og Benfey, 2018; Yang et al., 2018). Selv om et økende antall circRNAs blir identifisert, forblir deres funksjoner i planter og dyr generelt å bli studert. I tillegg til deres funksjoner som miRNA decoys, har circRNAs viktig translasjonspotensial, men ingen verktøy er tilgjengelige for å spesifikt forutsi translasjonsegenskapene til disse molekylene (Jakobi og Dieterich, 2019).
Det finnes Flere verktøy for prediksjon og identifisering av circRNAs, slik SOM CIRI (Gao et al., 2015), Jørgensen (Jørgensen et al., 2019), Jørgensen (Jørgensen et al., 2017), og circtools (Jakobi et al., 2018). Blant Dem Kan CircPro avsløre oversatte circrnaer ved å beregne en mulig poengsum for circrnaer basert på Cpc (Kong Et al ., 2007), som er et verktøy for å identifisere den åpne leserammen (ORF) i en gitt rekkefølge. Men fordi noen circRNAs ikke bruker startkodonet under oversettelse (Ingolia et al., 2011; Slav et al., 2013; Kearse og Wilusz, 2017; Spealman et al., 2018), ansette CPC kan filtrere ut noen virkelig oversatt circRNAs. I denne studien brukte Vi BASiNET (Ito et al., 2018), som er EN RNA-klassifiserer basert på maskinlæringsmetodene (random forest og J48-modellen). Den forvandler i utgangspunktet de gitte kodende Rna (positive data) og ikke-kodende rna (negative data) og representerer dem som komplekse nettverk; det trekker deretter ut de topologiske tiltakene til disse nettverkene og konstruerer en funksjonsvektor for å trene modellen som brukes til å klassifisere kodingskapasiteten til circrna. Med denne metoden unngås feilaktig filtrering av oversatte circRNAs som ikke er initiert AV AUG. I Tillegg Ribo-seq teknologi, som er basert på høy gjennomstrømning sekvensering for å overvåke RPFs (ribosomal beskyttet fragmenter)av transkripsjoner (Guttman et al., 2013; Brar and Weissman, 2015), kan brukes til å bestemme plasseringen av circRNAs som blir oversatt(Michel Og Baranov, 2013). For å identifisere kodingsevnen til circRNAs utviklet vi Verktøyet CircCode, som innebærer Et Python 3–basert rammeverk, og brukte CircCode for å undersøke oversettelsespotensialet til circRNAs fra mennesker og Arabidopsis thaliana. Vårt arbeid gir en rik ressurs for videre studier av funksjonene til circRNAs med kodingskapasitet.
Metoder
Sirkcode ble skrevet I Programmeringsspråket Python 3; Det bruker Trimmomatic (Bolger et al., 2014), bowtie (Langmead Og Salzberg, 2012), OG STAR (Dobin et al., 2013) for å filtrere rå Ribo-seq leser og kartlegge disse filtrerte leser til genomet. Kretskode identifiserer Deretter ribo-seq-lesetilordnede områder i kretsnaer som inneholder veikryss. Deretter sorteres kandidatkodede sekvenser i sirkrna basert på klassifiserere (J48-modell) i kodende Rna og ikke-kodende Rna ved BASiNET. Til slutt identifiseres korte peptider produsert ved oversettelse som potensielle kodende regioner av circrna. Hele Prosessen Med Sirkelkode består av fem trinn (Figur 1).
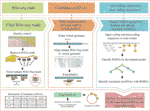
Figur 1 arbeidsflyten Til Sirkoden. Det øverste laget representerer inndatafilen som kreves for hvert Trinn I Sirkcode. Mellomlaget er delt inn i tre deler, og hver del representerer et annet operasjonsstadium. Fra venstre til høyre representerer den første delen filtreringen Av Ribo-seq-dataene; kvalitetskontrollen utføres Av Trimmomatic, og rRNA-lesingene fjernes av bowtie. Den andre delen representerer trinnene som brukes til å produsere det virtuelle genomet og justere den filtrerte leser til det virtuelle genomet MED STJERNE. Den siste delen representerer identifisering av oversatte circRNAs ved maskinlæring. Bunnlaget representerer det siste trinnet som brukes til å forutsi peptidene oversatt fra circrna og de endelige resultatene, inkludert informasjon om oversatte circrna og deres oversettelsesprodukter.
Filtrering Av Ribosomale Profileringsdata
for det første fjernes fragmenter og adaptere av lav kvalitet i Ribo-Seq-avlesningene med Trimmomatic med standardparametrene for å oppnå rene Ribo-seq-avlesninger. For det andre er disse rene Ribo-seq-lesene kartlagt til et rrna-bibliotek for å fjerne leser avledet fra rRNA ved hjelp av bowtie. Fordi leselengdene Til Ribo-seq er relativt korte (vanligvis mindre enn 50 bp), er det mulig for en les å matche flere regioner. I dette tilfellet er det vanskelig å avgjøre hvilken region en bestemt les tilsvarer. For å unngå dette, de rene Ribo-seq leser er kartlagt til genomet av en art av interesse, og leser som ikke er perfekt justert til genomet regnes som den endelige unike Ribo-seq leser.
Montering Av Virtuelle Genomer
Sirkrnaer vises vanligvis som ringformede molekyler i eukaryoter, og de kan identifiseres basert på deres spleisekryss. Sekvensene av circRNAs i fasta-filen er imidlertid ofte i lineær form. I teorien indikerer resultatet at krysset er mellom 5 ‘terminal nukleotid og 3’ terminal nukleotid, selv om krysset og sekvensen nær krysset ikke kan ses direkte, og dermed justere Ribo-seq leser til circRNA sekvenser, inkludert veikryss, på en grei måte.
Sirkelkode forbinder sekvensen av hver circRNA i tandem slik at krysset for hver er midt i den nybygde sekvensen. Vi separerte også hver serieenhet med 100 n nukleotider for å unngå forvirring ved sekvensjusteringstrinnet (lengden på hver RPF er mindre enn 50 bp). Til slutt fikk vi et virtuelt genom som bare består av kandidat circRNAs i tandem separert med 100 Ns. Fordi CircCode fokuserer bare på justering Mellom Ribo-seq leser og circRNA-sekvenser, kan vi undersøke kodingspotensialet til circRNAs ved å kartlegge Ribo-seq leser til dette virtuelle genomet, som kan spare en stor mengde beregningstid (det virtuelle genomet er mye mindre enn hele genomet) og øke nøyaktigheten (ved å unngå forstyrrelser mellom oppstrøms og nedstrøms sekvenssammenligninger av circRNAs).
Bestemmelse Av Ribo-seq-Lesekartet Region på ET Veikryss (RMRJ) av circRNAs
den endelige unike Ribo-seq-lesingen er kartlagt til et tidligere opprettet virtuelt genom ved HJELP AV STJERNE. Fordi hver tandem circRNA-enhet ble separert med 100 n baser før det virtuelle genomet ble produsert, ble den største intronlengden satt til ikke å overstige 10 baser med parameteren » – alignIntronMax 10.»Denne parameteren eliminerer enhver interaksjon mellom forskjellige circRNAs i sekvensjusteringen. I det andre trinnet av virtual genome produksjon, CircCode lagrer posisjons veikryss informasjon for hver circRNA i det virtuelle genomet. Hvis Ribo-seq lese-kartlagt region i det virtuelle genomet omfatter krysset av circRNA, og antall kartlagt Ribo-seq leser på junction (NMJ) er større enn 3, Ribo-seq leser-kartlagt region på krysset av circRNAs kan betraktes som EN RMRJ, som avslører et grovt oversatt segment av circRNAs nær krysset området.
Opplæring Av Modell Og Klassifisering Av RMRJs
Selv Om RMRJs kan utgjøre kraftig bevis på oversettelse, er det fortsatt noen mangler i denne metoden. Fordi lengden på lesingen av det ribosomale kartet er kort, kan en lesning sammenlignes med feil posisjon. Derfor er det ikke overbevisende å bare vurdere regionen som Dekkes Av Ribo-seq leser som den oversatte regionen. Til dette formål brukes maskinlæringsmetoden til å identifisere KODINGSEVNEN TIL RMRJ. For Det første trekker Sirkoden ut kodende Rna (positive data) og ikke-kodende rna (negative data) fra en art av interesse og bruker dem til modellopplæring ved hjelp av forskjellen i funksjonsvektorer mellom koding og ikke-kodende Rna. CircCode bruker deretter trent modell for å klassifisere RMRJs oppnådd i forrige trinn Av BASiNET. HVIS RMRJ av en circRNA er anerkjent som kodende RNA, kan denne circRNA identifiseres som en oversatt circRNA.
Prediksjon Av Oversatte Peptider Av RMRJs
som uttrykk for circrna i organismer er lav, Ribo-seq data viser ikke nøyaktig 3-nt periodisitet klart i tilfelle av færre RPFs. Derfor er det vanskelig å fastslå nøyaktig oversettelse start stedet for en oversatt circRNA. På grunn av tilstedeværelsen av et stoppkodon i Noen RMRJs og fordi startkodonet er vanskelig å bestemme, er metoden for å finne EN ORF basert på et startkodon og et stoppkodon ikke mulig.
for å fastslå den sanne oversettelse regioner av disse circRNAs og generere den endelige oversettelse produkt, FragGeneScan (Rho et al., 2010), som kan forutsi proteinkodende regioner i fragmenterte gener og gener med rammeskift, brukes til å bestemme de oversatte peptidene produsert av circrna.
for å unngå den besværlige kjøringsprosessen, kan alle modellene kalles av et skallskript; brukeren kan bare fylle ut den oppgitte konfigurasjonsfilen og legge den inn i skriptet, og hele prosessen for å forutsi de oversatte circRNAs vil da bli kjørt. I tillegg Kan Sirkoden kjøres separat, trinnvis, slik at brukeren kan justere parametrene midt i prosedyren og se resultatene av hvert trinn som ønsket.
Resultater Og Diskusjon
Etter testing på flere datamaskiner ble Det funnet At Sirkelkoden kjørte med de nødvendige avhengighetene installert. For å teste Ytelsen Til CircCode, vi brukte data for mennesker og A. thaliana å forutsi circRNAs med oversettelse potensial. Resultatene ble sammenlignet med circRNAs som har blitt verifisert eksperimentelt som bekreftelse. Deretter testet vi verdien false discovery rate (fdr) av CircCode ytterligere. Vi brukte GenRGenS (Ponty et al., 2006) for å generere et datasett for testing basert på kjente oversatte circRNAs og bekreftet AT FDR-verdien var innenfor et akseptabelt område og på et lavt nivå. Til slutt evaluerte vi effekten av forskjellige sekvenseringsdybder Av Ribo-seq-data på Sirkcode-spådommer og sammenlignet Sirkcode med annen programvare.
Oversatt circRNAs I Mennesker Og A. thaliana
for å bruke CircCode-verktøyet til ekte data, lastet vi først ned filene, inkludert det menneskelige referansegenomet GRCh38, genome annotation og human rRNA, Fra Ensembl. For a. thaliana ble referansegenomene (TAIR10), genom-annotasjonsfiler og tilhørende rrna-sekvenser alle lastet ned fra Ensembleplanter. Ribo-seq-dataene for mennesker og a. thaliana ble lastet ned Fra RPFdb( tiltredelsesnumre: GSE96643, GSE81295, GSE88794) (Hsu et al., 2016; Willems et al., 2017), og alle kandidatene circRNAs fra human og A. thaliana ble lastet ned fra CIRCPedia v2 (Dong et al., 2018) Og PlantcircBase, henholdsvis (Chu et al., 2017). Til slutt identifiserte vi 3 610 oversatte circrna fra mennesker og 1 569 oversatte circrna fra a. thaliana ved Hjelp Av CircCode (Supplerende Data 1).
Funksjonell Berikelse Av Menneske og a. thaliana circRNAs Med Kodingspotensial
Ved Hjelp Av CircCode-resultatene for menneske og a. thaliana, det elektroniske verktøyet KOBAS 3.0 (Wu et al ., 2006) ble ansatt for å annotere disse oversatte kretsene basert på deres overordnede gener. Videre utførte VI GO (Gene Ontology) funksjonell analyse og Kegg (Kyoto Encyclopedia Of Genes and Genomes) anrikningsanalyse for disse oversatte circRNAs ved Hjelp Av r-pakken clusterProfiler (Yu et al., 2012).
kegg-resultatene viste at de humane sirkrnaene ble beriket i proteinbehandling i endoplasmatisk retikulumbane, karbonmetabolismebane og RNA-transportvei. GO analyse indikerte deltakelse av menneskelige oversatt circRNAs i regulering av molekylbinding, ATPase aktivitet, og ANDRE rna spleising-relaterte biologiske prosesser. I tillegg er de oversatte circRNAs Av A. thaliana beriket i veier relatert til stressmotstand, noe som tyder på at de spiller viktige roller i denne prosessen (Supplerende Data 2).
Nøyaktighetstest For Sirkkode
for å undersøke Nøyaktigheten Av Sirkoden ble testsekvenser generert Av GenRGenS, som bruker den skjulte Markov-modellen til å produsere sekvenser som har samme sekvensegenskaper (for eksempel frekvensene av forskjellige nukleotider, forskjellige kodoner Og forskjellige nukleotider ved starten av sekvensen), brukt.
for denne studien brukte vi tidligere publiserte menneskelige oversatte circRNAs (Yang et Al., 2017) som input For GenRGenS og genererte 10.000 sekvenser for å teste Sirkcode. Vi gjentok testen 10 ganger, og i gjennomsnitt ble 27 oversatte circRNAs spådd hver gang. FDR-verdien ble beregnet til å være 0,0027, som er mye mindre enn 0,05, noe som indikerer at de forventede resultatene er troverdige.
i tillegg sammenlignet vi de oversatte circrna fra mennesker som identifisert av CircCode med verifiserte polysomassosierte circRNA data (Yang et al., 2017). Blant dem ble 60% av sirkrna identifisert Med Sirkkode (Supplerende Data 3).
Påvirkning Av Ribo-seq Datasekvenseringsdybde
for å undersøke virkningen av sekvenseringsdybden Av Ribo-seq-data på Sirkelkodeidentifikasjonsresultatene, testet vi først effekten av sekvenseringsdybde på antall oversatte sirkrnaer (Figur 2a). Når sekvenseringsdybden var lav, det anslåtte antall oversatte circRNAs var lav, og antall oversatte circRNAs økt med økende sekvensering dybde. Antallet oversatte circRNAs ble stabilt når sekvenseringsdybden nådde ikke mindre enn 10× lineær transkripsjonsdekning.

Figur 2 (A) Effekt Av Ribo-seq datasekvenseringsdybde på det anslåtte antall oversatte sirkrna. (B) effekten av junction read number (JRN) på Sirkelkodefølsomhet ved forskjellige sekvenseringsdybder.
For Det Andre ble OGSÅ PÅVIRKNING AV NMJ på følsomhet ved forskjellige sekvenseringsdybder vurdert (Figur 2b). RESULTATENE viste at NMJ hadde mindre innvirkning på sensitiviteten etter hvert som sekvenseringsdybden økte. Sirkoden hadde også høyere følsomhet ved Bruk Av Ribo-seq-data med høyere sekvenseringsdybde.
Sammenligning Av Kretskode Med Andre Verktøy
for å sammenligne Kretskode med andre verktøy, for eksempel CircPro, ble Det samme settet Med Ribo-seq-data (SRR3495999) Fra A. thaliana brukt til å identifisere oversatte kretskoder ved hjelp av seks prosessorer, med 16 gigabyte RAM. CircPro identifiserte 44 oversatte circRNAs på 13 minutter, Mens CircCode identifiserte 76 oversatte circRNAs på 20 minutter. Dermed Er Sirkoden mer følsom enn CircPro på samme maskinvarenivå, men det tar mer tid. CircPro er konsis og mindre tidkrevende Enn CircCode, Men CircCode kan identifisere flere circRNAs med koding evne Enn CircPro.
Konklusjoner
Sirkrna spiller en viktig rolle i biologi, og det er avgjørende å nøyaktig identifisere sirkrna med kodingsevne for senere forskning. Basert På Python 3 utviklet Vi CircCode, et brukervennlig kommandolinjeverktøy som har høy følsomhet for å identifisere oversatte circRNAs fra Ribo-Seq leser med høy nøyaktighet. Sirkelkode viser god ytelse i både planter og dyr. Fremtidig arbeid vil legge nedstrøms karakteranalyse Til CircCode ved å visualisere hvert trinn i prosessen og optimalisere nøyaktigheten av prediksjonen.
Tilgjengelighet Og Krav
CircCode er tilgjengelig på https://github.com/PSSUN/CircCode; operativsystem(er): Linux, programmeringsspråk: Python 3 Og R; andre krav: bedtools (versjon 2.20.0 eller nyere), bowtie, STAR, Python 3 pakker (Biopython, Pandas, rpy2), R-pakker (BASiNET, Bistrings). Installasjonspakkene for all nødvendig programvare er tilgjengelig på CircCode hjemmeside. Brukere trenger ikke å laste dem ned individuelt. Den CircCode hjemmesiden gir også detaljerte brukerhåndbøker for referanse. Verktøyet er fritt tilgjengelig. Det er ingen restriksjoner på bruk av nonacademics.
Datatilgjengelighetserklæring
alle relevante data er i manuskriptet og Dets Støtteinformasjonsfiler.
Forfatterbidrag
Konseptualisering: PS, GL. Data Curation: PS, GL. Formell Analyse: PS, GL. Skrive-Originalt Utkast: PS, GL. Skriving-Gjennomgang OG Redigering: PS, GL.
Finansiering
dette arbeidet ble støttet av tilskudd Fra National Natural Science Foundation Of China (grant nos. 31770333, 31370329, og 11631012), Programmet For New Century Gode Talenter I Universitetet (NCET-12-0896), Og De Grunnleggende Forskningsmidler For De Sentrale Universiteter (nr. GK201403004). Finansieringsbyråene hadde ingen rolle i studien, utformingen, datainnsamlingen og analysen, beslutningen om å publisere eller utarbeidelsen av manuskriptet. Finansiørene hadde ingen rolle i studiedesign, datainnsamling og analyse, beslutning om publisering eller utarbeidelse av manuskriptet.
Interessekonflikter
forfatterne erklærer at forskningen ble utført i fravær av kommersielle eller økonomiske forhold som kan tolkes som en potensiell interessekonflikt.
Supplerende Materiale
Supplerende Materiale for denne artikkelen finner du online på: https://www.frontiersin.org/articles/10.3389/fgene.2019.00981/full#supplementary-material
Supplerende Data 1 / sekvensen av den spådde oversatt circRNA og kort peptid.
Supplerende Data 2 / GO berikelse og kegg berikelse resultater for mennesker Og Arabidopsis thaliana.
Supplerende Data 3 | Sammenligning av anslåtte oversatte circRNAs med validerte oversatte circRNAs.
Bolger, A. M., Lohse, M., Usadel, B. (2014). Trimmomatic: en fleksibel trimmer for illumina sekvensdata. Bioinformatikk 30, 2114-2120. doi: 10.1093/bioinformatikk | btu170
PubMed Abstrakt / CrossRef Fulltekst / Google Scholar
Brar, G. A., Weissman, J. S. (2015). Ribosom profilering avslører hva, når, hvor og hvordan av proteinsyntese. Nat. Pastor Mol. Celle Biol. 16, 651–664. doi: 10.1038 / nrm4069
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Chen, L.-L., Yang, L. (2015). Regulering av circRNA biogenesis. RNA Biol. 12, 381–388. doi: 10.1080/15476286.2015.1020271
PubMed Abstrakt / Fulltekst / Google Scholar
Chu, Q., Zhang, X., Zhu, X., Liu, C., Mao, L., Ye, C., Et al. (2017). PlantcircBase: en database for anlegg sirkul RNAs. Mol. Plante 10, 1126-1128. doi: 10.1016 / j.molp.2017.03.003
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Schlesinger, F., Drenkow, J., Zaleski, C., Jha, S., et al. (2013). STJERNE: ultrafast universal rna-seq aligner. Bioinformatikk 29, 15-21. doi: 10.1093 / bioinformatikk / bts635
PubMed Abstrakt | CrossRef Fulltekst / Google Scholar
Dong, R., Ma, X.-K., Chen, L.-L., Yang, L. (2019). «Genom-wide annotasjon av circRNAs og deres alternative back-skjøting / skjøting Med CIRCexplorer Rørledning,» I Epitranscriptomics. Eds. Wajapeyee, N., Gupta, R. (New York, NY: Springer New York), 137-149. doi: 10.1007/978-1-4939-8808-2_10
Full Text | Google Scholar
Dong, R., Ma, X.-K., Li, G.-W., Yang, L. (2018). CIRCpedia v2: en oppdatert database for omfattende sirkulær rna-merknad og uttrykkssammenligning. Genomics Proteomics Bioinf. 16, 226–233. doi: 10.1016 / j. gpb.2018.08.001
CrossRef Fulltekst | Google Scholar
Gao, Y., Wang, J., Zhao, F. (2015). CIRI: en effektiv og objektiv algoritme for de novo sirkulær rna-identifikasjon. Genom Biol. 16, 4. doi: 10.1186 / s13059-014-0571-3
PubMed Abstrakt / Fulltekst / Google Scholar
Guttman, M., Russell, P., Ingolia, N. T., Weissman, J. S., Lander ,E. S. (2013). Ribosomprofilering gir bevis på at store ikke-kodende Rna ikke koder for proteiner. Celle 154, 240-251. doi: 10.1016 / j.celle.2013.06.009
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Hsu, P. Y., Benfey ,P. N. (2018). Liten, men mektig: funksjonelle peptider kodet av små ORFs i planter. PROTEOMIKK 18, 1700038. doi: 10.1002 / pmic.201700038
CrossRef Fulltekst | Google Scholar
Hsu, P. Y., Calviello, L., Wu, H.-Y. L., Li, F.-W., Rothfels, C. J., Ohler, U., et al. (2016). Super-oppløsning ribosom profilering avslører uanmeldte oversettelse hendelser I Arabidopsis. Proc. Natl. Acad. Sci. 113, E7126-E7135. doi: 10.1073 / pnas.1614788113
Kryssref Fulltekst | Google Scholar
Ingolia, N. T., Lareau, L. F., Weissman, J. S. (2011). Ribosomprofilering av musembryonale stamceller avslører kompleksiteten og dynamikken til pattedyrsproteomer. Celle 147, 789-802. doi: 10.1016 / j.celle.2011.10.002
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Ito, E. A., Katahira, I., Vicente, F. F., da, r., Pereira, L. F. P., Lopes, F. M. (2018). BASiNET-Biologiske Sekvenser Nettverk: en case studie på koding Og ikke-koding Rna identifikasjon. Nukleinsyrer Res. 46, e96-e96. doi: 10.1093/nar/gky462
PubMed Abstract | CrossRef Full Text | Google Scholar
Jakobi, T., Dieterich, C. (2019). Computational approaches for circular RNA analysis. Wiley Interdiscip. Rev. RNA,10 (3), e1528. doi: 10.1002/wrna.1528
PubMed Abstract | CrossRef Full Text | Google Scholar
Jakobi, T., Uvarovskii, A., Dieterich, C. (2018). circtools—a one-stop software solution for circular RNA research. Bioinformatics 35 (13), 2326–2328. doi: 10.1093 / bioinformatikk / bty948
CrossRef Fulltekst | Google Scholar
Kearse, M. G., Wilusz ,J. E. (2017). Non-AUG oversettelse: en ny start for proteinsyntese i eukaryoter. Gener Dev. 31, 1717–1731. doi: 10.1101 / gad.305250.117
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Kong, L., Zhang, Y., Ye, Z.-Q., Liu, X.-Q., Zhao, S.-Q., Wei, L., et al. (2007). Cpc: vurder proteinkodingspotensialet til transkripsjoner ved hjelp av sekvensfunksjoner og støttevektormaskin. Nukleinsyrer Res. 35, W345-W349. doi: 10.1093 / nar / gkm391
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Langmead, B., Salzberg, S. L. (2012). Rask gapped-les justering Med Bowtie 2. Nat. Metoder 9, 357-359. doi: 10.1038 / nmeth.1923
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Meng, X., Chen, Q., Zhang, P., Chen, M. (2017). CircPro: et integrert verktøy for identifisering av circrna med proteinkodingspotensial. Bioinformatikk 33, 3314-3316. doi: 10.1093 / bioinformatikk / btx446
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Michel, A. M., Baranov, P. V. (2013). Ribosomprofilering: En Hi-Def-skjerm for proteinsyntese i genom-bred skala: ribosomprofilering. Wiley Interdiscip. Rna 4, 473-490. doi: 10.1002 / wrna.1172
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Jens, M., Ashwal-Fluss, R., Stottmeister, C., Ruhe, L., Et al. (2017). Oversettelse Av CircRNAs. Mol. Celle 66, 9-21.e7. doi: 10.1016 / j.molcel.2017.02.021
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Ponty, Y., Termier, M., Denise ,A. (2006). GenRGenS: programvare for å generere tilfeldige genomiske sekvenser og strukturer. Bioinformatikk 22, 1534-1535. doi: 10.1093 / bioinformatikk / btl113
PubMed Abstrakt | CrossRef Fulltekst / Google Scholar
Rho, M., Tang, H., Ye, Y. (2010). FragGeneScan: forutsi gener i korte og feilutsatte leser. Nukleinsyrer Res. 38, e191-e191. doi: 10.1093/forskerforbundet/gkq747
PubMed Abstrakt | CrossRef Full Tekst | Google Scholar
Slavoff, S. A., Mitchell, A. J., Schwaid, A. G., Cabili, M. N., Ma, J., Levin, J. Z., et al. (2013). Peptidomisk oppdagelse av korte åpne leserammekodede peptider i humane celler. Nat. Chem. Biol. 9, 59–64. doi: 10.1038 / nchembio.1120
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Spealman, P., Naik, A. W., May, G. E., Kuersten, S., Freeberg, L., Murphy, R. F., Et al. (2018). Konserverte ikke-AUG uORFs avslørt av en ny regresjonsanalyse av ribosomprofileringsdata. Genom Res. 28, 214-222. doi: 10.1101 / gr.221507.117
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Vicens, Q., Westhof, E. (2014). Biogenese av sirkulære Rna. Celle 159, 13-14. doi: 10.1016 / j.celle.2014.09.005
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Willems, P., Ndah, E., Jonckheere, V., Stael, S., Sticker, A., Martens, L., et al. (2017). N-terminal proteomics assistert profilering av uutforsket oversettelse initiering landskapet I Arabidopsis thaliana. Mol. Celle. Proteomikk 16, 1064-1080. doi: 10.1074 / mcp.M116. 066662
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Wu, J., Mao, X., Cai, T., Luo, J., Wei, L. (2006). KOBAS server: en nettbasert plattform for automatisert annotering og veiidentifikasjon. Nukleinsyrer Res. 34, W720-W724. doi: 10.1093 / nar / gkl167
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Yang, L., Fu, J., Zhou, Y. (2018). Sirkulære Rna og Deres Fremvoksende Roller I Immunregulering. Front. Immunol. 9, 2977. doi: 10.3389 / fimmu.2018.02977
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
J., J., J., J., J., J., J., J., J., J., J., j., et al. (2017). Omfattende oversettelse av sirkul Rna drevet Av N6-metyladenosin. Celle Res. 27, 626-641. doi: 10.1038 / cr.2017.31
PubMed Abstrakt | CrossRef Fulltekst | Google Scholar
Yu, G., Wang, L.-G. ,Han, Y., Han, Q.-Y. (2012). clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS J. Integr. Biol. 16, 284–287. doi: 10.1089/omi.2011.0118
CrossRef Full Text | Google Scholar