Lundi 21 septembre 2020
Même dans le domaine de la génomique où de nouvelles percées se produisent tous les quelques mois, la réalisation du tout premier autosome humain entièrement séquencé est une réalisation capitale. Très précis, pas de lacunes, pas de jointures erronées – juste le chromosome 8 dans toute sa splendeur. C’est un exploit remarquable et nous sommes honorés que les lectures PacBio HiFi aient joué un rôle central pour y parvenir.
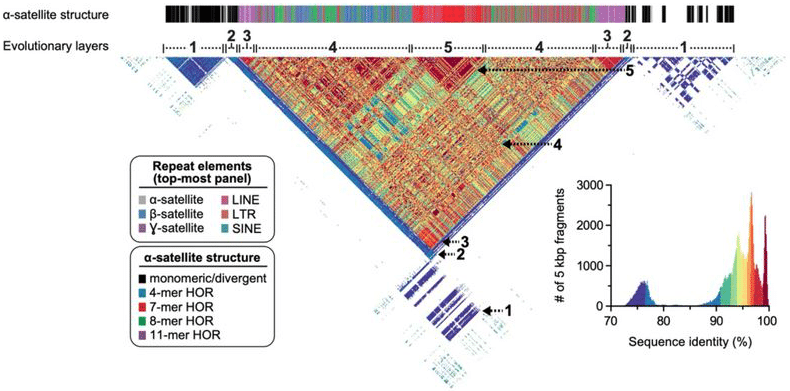
La séquence complète des centromères du chromosome 8 montre une diversité de répétitions satellites et d’autres répétitions génomiques abondantes, maintenant avec une résolution de base presque parfaite d’un bout à l’autre. G et coll. (2020)
Ce travail est décrit dans une préimpression publiée récemment sur bioRxiv par l’auteur principal Glennis Logsdon (@glennis_logsdon), l’auteur principal Evan Eichler et leurs collaborateurs du Consortium Télomère à télomère (T2T). Il fait partie de l’initiative T2T plus large visant à séquencer et à assembler le premier génome humain véritablement complet et fait suite à la libération antérieure du chromosome X entièrement séquencé.
« Depuis l’annonce du séquençage du génome humain il y a 20 ans, les chromosomes humains sont restés inachevés en raison de grandes régions de répétitions très identiques situées dans les centromères, de la duplication segmentaire et des bras courts acrocentriques des chromosomes », notent les auteurs. » L’avènement des technologies de séquençage à lecture longue et des algorithmes associés a permis pour la première fois d’assembler systématiquement ces régions à partir d’ADN natif. »
Le chromosome 8 constituait une cible attrayante pour le premier autosome du T2T en raison de son centromère gérable (estimé précédemment à 1,5 Mo à 2,2 Mo de long). Mais le chromosome abrite également « l’une des régions les plus structurellement dynamiques du génome humain — le groupe de gènes β-défensine situé en 8p23.1 — ainsi qu’un néocentromère situé en 8q21.2, qui ont été en grande partie non résolus au cours des 20 dernières années « , écrivent les scientifiques. L’amas de β-défensine joue un rôle clé dans l’immunité innée et la variation structurelle dans cette région a longtemps été impliquée dans la maladie humaine.
Le nouvel ensemble, qui répond aux cinq lacunes auparavant insolubles du génome de référence humain, a été construit avec une méthode intelligente utilisant plusieurs ensembles de données, y compris des lectures longues précises: « Plus de la moitié des données PacBio HiFi sont contenues dans des lectures supérieures à 17,8 kbp, avec une précision médiane supérieure à 99,9%. »Après une étape d’échafaudage basée sur des lectures de nanopores Oxford, des contigs assemblés à partir de lectures PacBio HiFi ont été échangés pour fournir la résolution de la paire de base. « Nous avons amélioré la précision des paires de bases des échafaudages de séquences en remplaçant la séquence ONT brute par plusieurs contigs PacBio HiFi concordants », rapporte l’équipe.
La séquence chr8 complète s’affiche à 146 Mo et comprend plus de 3 Mo manquants dans GRCh38. Comme Logsdon et coll. écrivez: « Le résultat est un assemblage de chromosomes entiers avec une précision estimée de la paire de bases supérieure à 99,99%. »
Les scientifiques ont également abordé ce groupe de gènes β-défensine persnickety, « que nous avons résolu en un seul locus de 7,06 Mbp – nettement plus grand que la région de 4,56 Mbp du génome de référence humain actuel », notent-ils. Presque toutes ces données de séquence — 99,9934%, pour être précis – provenaient de lectures HiFi. Le centromère complet, quant à lui, représentait 2,08 Mo.
Avec ce bel assemblage en main, l’équipe de T2T l’a sorti pour faire un tour. Tout d’abord, ils l’ont validé avec une foule d’outils orthogonaux, tels que le mappage optique. Ensuite, ils ont généré des données HiFi pour les orthologues du chromosome 8 chez le chimpanzé, le macaque et l’orang-outan afin de comparer les données de séquence et de reconstruire l’histoire évolutive de l’autosome humain. « Des analyses comparatives et phylogénétiques montrent que la structure du satellite α d’ordre supérieur a évolué spécifiquement chez l’ancêtre des grands singes, et que la région centromérique a évolué avec une symétrie en couches », écrit l’équipe. « Nous estimons que le taux de mutation de l’ADN satellite centromérique est accéléré d’au moins 2.2 fois, et cette accélération s’étend au-delà du satellite α d’ordre supérieur dans la séquence flanquante. »
Enfin, les chercheurs ont effectué une analyse des transcriptions complètes produites avec la méthode Iso-Seq. Ce processus a permis d’identifier « 61 loci codants pour les protéines et 33 loci non codants qui correspondent mieux à cette séquence du chromosome 8 terminée qu’à GRCh38, y compris la découverte de nouveaux gènes cartographiant les régions polymorphes du nombre de copies », rapportent-ils. Douze de ces nouveaux gènes ont été découverts uniquement dans ce locus délicat de la β-défensine.
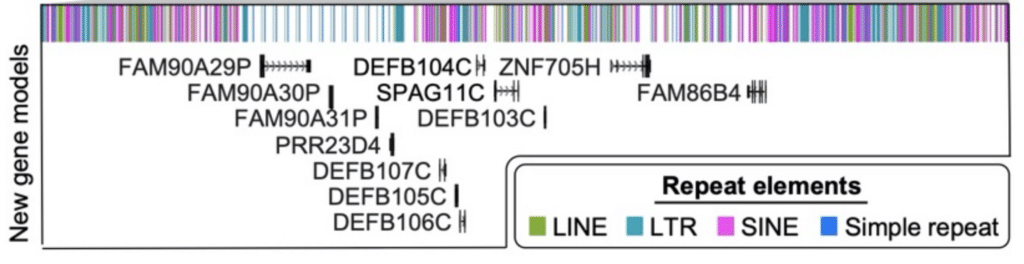
Une combinaison d’assemblage du génome HiFi et d’annotation de l’ARN avec des données Iso-Seq a ajouté ces 12 nouveaux gènes à la région de la β-défensine (DEFB) du chromosome 8. G et coll. (2020)
Pour beaucoup d’entre nous dans la communauté de la génomique, cet article représente bien plus que la séquence d’un seul chromosome humain. C’est une déclaration sur ce que la science peut accomplir maintenant, et où cela peut nous mener dans les années à venir. Comme l’ont résumé les auteurs: « Maintenant que des régions complexes comme celles-ci peuvent être séquencées et assemblées, il sera important d’étendre ces analyses à d’autres centromères, à de multiples individus et à d’autres espèces pour comprendre leur impact complet sur la variation et l’évolution génétiques. »
Vous pouvez entendre plus de détails de Logsdon directement lors d’une conférence en ligne gratuite co-organisée par le Consortium T2T et le Human Pangenome Reference Consortium (HPRC) les 22 et 23 septembre. Les conférenciers offriront de nouvelles perspectives sur le chromosome 8 et rendront compte des progrès du T2T vers un assemblage complet du génome humain. Lors du même événement, le CRHPS présentera ses efforts complémentaires pour séquencer des centaines de génomes humains à haute qualité. Les présentateurs comprennent : Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike Schatz (@mike_schatz), Heng Li (@lh3lh3) et bien d’autres. Nous serons collés à nos ordinateurs pour cela et nous espérons que vous aurez également la chance de participer!