mandag den 21.September 2020
selv inden for genomik, hvor nye gennembrud forekommer hvert par måneder, er færdiggørelsen af det første nogensinde fuldt sekventerede menneskelige autosom en vigtig præstation. Meget præcis, ingen huller, ingen mis-joints — bare kromosom 8 i al sin herlighed. Det er en bemærkelsesværdig bedrift, og vi er beærede over, at PacBio HiFi reads spillede en central rolle i at hjælpe med at opnå det.
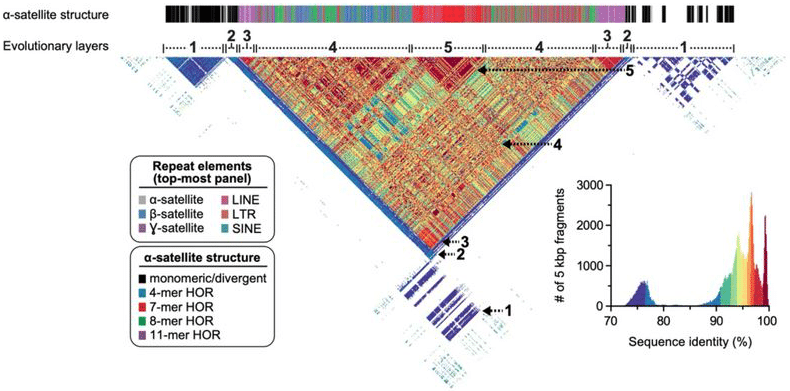
den komplette centromere sekvens af kromosom 8 viser en mangfoldighed af satellit gentagelser og andre rigelige genomiske gentagelser, nu med næsten perfekt base-niveau opløsning fra ende til ende. Logsdon, G et al. (2020)
dette arbejde er beskrevet i et preprint, der for nylig blev sendt til telomer fra hovedforfatter Glennis Logsdon (@glennis_logsdon), seniorforfatter Evan Eichler og deres samarbejdspartnere i telomer-to-telomer (T2T) konsortiet. Det er en del af det bredere T2T-initiativ til at sekvensere og samle det første virkelig komplette menneskelige genom og følger den tidligere frigivelse af det fuldt sekventerede kromosom.
“siden meddelelsen om sekventering af det menneskelige genom for 20 år siden har menneskelige kromosomer været ufærdige på grund af store regioner med meget identiske gentagelser placeret inden for centromerer, segmental duplikering og de akrocentriske korte arme af kromosomer,” bemærker forfatterne. “Fremkomsten af langlæste sekventeringsteknologier og tilknyttede algoritmer har nu gjort det muligt systematisk at samle disse regioner fra nativt DNA for første gang.”
kromosom 8 gjorde et attraktivt mål for T2T ‘ s første autosome på grund af dets håndterbare centromere (tidligere estimeret til 1, 5 Mb til 2, 2 Mb lang). Men kromosomet er også hjemsted for “en af de mest strukturelt dynamiske regioner i det menneskelige genom—genklyngenen, der er placeret på 8p23.1-samt en neocentromere placeret på 8k21.2, som stort set har været uløst i de sidste 20 år,” skriver forskerne. Kristian-defensin-klyngen spiller en nøglerolle i medfødt immunitet, og strukturel variation i denne region har længe været impliceret i menneskers sygdom.
den nye samling, der adresserer alle fem af de tidligere uhåndterlige huller i det humane referencegenom, blev bygget med en smart metode ved hjælp af flere datasæt, herunder nøjagtige lange læser: “mere end halvdelen af PacBio HiFi-dataene er indeholdt i læser større end 17,8 kbp, med en median nøjagtighed på over 99,9%.”Efter et stilladstrin baseret på Nanopore læser, blev contigs samlet fra PacBio HiFi læser byttet ind for at give basisparopløsningen. “Vi forbedrede baseparets nøjagtighed af sekvensstilladserne ved at erstatte den rå ONT-sekvens med flere konkordante PacBio HiFi contigs,” rapporterer teamet.
den komplette chr8 sekvens ure i på 146 Mb og omfatter mere end 3 Mb mangler fra GRCh38. Som Logsdon et al. skriv, ” resultatet er en hel-kromosom-samling med en estimeret baseparnøjagtighed på over 99,99%.”
forskerne tacklede også den persnickety Krishn-defensin – genklynge,” som vi løste til et enkelt 7.06 Mbp—locus-væsentligt større end 4.56 Mbp-regionen i det nuværende humane referencegenom, ” bemærker de. Næsten alle disse sekvensdata-99.9934% af dem, for at være præcise-kom fra HiFi læser. Den komplette centromere tegnede sig i mellemtiden for 2, 08 Mb.
med denne smukke samling i hånden tog T2T-teamet det ud for et spin. Først validerede de det med en række ortogonale værktøjer, såsom optisk kortlægning. Dernæst genererede De HiFi-data for kromosom 8-ortologerne i chimpanse, makak og orangutang for at sammenligne sekvensdataene og rekonstruere den menneskelige autosoms evolutionære historie. “Komparative og fylogenetiske analyser viser, at den højere orden af den højere orden udviklede sig specifikt i Den Store ape-forfader, og den centromere region udviklede sig med en lagdelt symmetri,” skriver holdet. “Vi vurderer, at mutationshastigheden for centromerisk satellit-DNA accelereres mindst 2.2-fold, og denne acceleration strækker sig ud over den højere orden af den højere orden i den flankerende sekvens.”
endelig udførte forskerne en analyse af transkripter i fuld længde produceret med Iso-Sekv-metoden. Denne proces identificerede ” 61 proteinkodende og 33 ikke-kodende loci, der kortlægger bedre til denne færdige kromosom 8-sekvens end til GRCh38, herunder opdagelsen af nye gener kortlægning for at kopiere antal polymorfe regioner,” rapporterer de. Tolv af disse nye gener blev afdækket i det vanskelige Krishna-defensin locus alene.
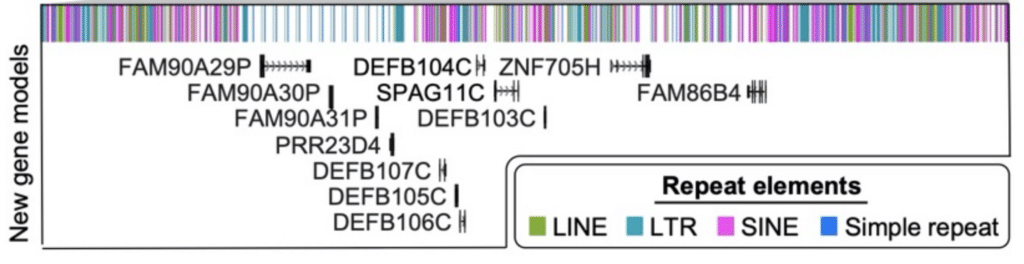
en kombination af HiFi-genomsamling og RNA-annotation med Iso-Sekv-data tilføjede disse 12 nye gener til regionen af kromosom 8. Logsdon, G et al. (2020)
for så mange af os i det genomiske samfund repræsenterer dette papir langt mere end sekvensen af et enkelt humant kromosom. Det er en erklæring om, hvad videnskaben kan udrette nu, og hvor det kan føre os i de kommende år. Som forfatterne opsummerede: “Nu hvor komplekse regioner som disse kan sekventeres og samles, vil det være vigtigt at udvide disse analyser til andre centromerer, flere individer og yderligere arter for at forstå deres fulde indflydelse med hensyn til genetisk variation og evolution.”
du kan høre flere detaljer fra Logsdon direkte på en gratis online konference, der er vært for T2T-konsortiet og Human Pangenome Reference Consortium (HPRC) den 22. / 23. September. Højttalere vil tilbyde ny indsigt i kromosom 8 og rapportere om yderligere T2T-fremskridt mod en komplet human genomsamling. Ved samme begivenhed vil HPRC præsentere sin komplementære indsats for at sekvensere hundredvis af menneskelige genomer til høj kvalitet. Præsentanter inkluderer: Karen Miga (@khmiga), Eric Green (@NHGRI_Director) Adam Phillippy (@aphillippy), Sergey Koren (@sergekoren), Sergey Nurk (@sergeynurk), Valerie Schneider (@dnadiver), Tina Graves-Lindsay, Arang Rhie (@ArangRhie), Mitchell R. Vollger (@mrvollger), Erich Jarvis (@erichjarvis), Mark Chaisson (@mjpchaisson), Mike schat (@Mike_schat), Heng Li (@Lh3lh3) og mange flere. Vi bliver limet til vores computere for det, og vi håber, at du også har en chance for at deltage!